第 4 章 GB2随机突变噬菌体文库的构建及淘选.docx
《第 4 章 GB2随机突变噬菌体文库的构建及淘选.docx》由会员分享,可在线阅读,更多相关《第 4 章 GB2随机突变噬菌体文库的构建及淘选.docx(20页珍藏版)》请在冰豆网上搜索。
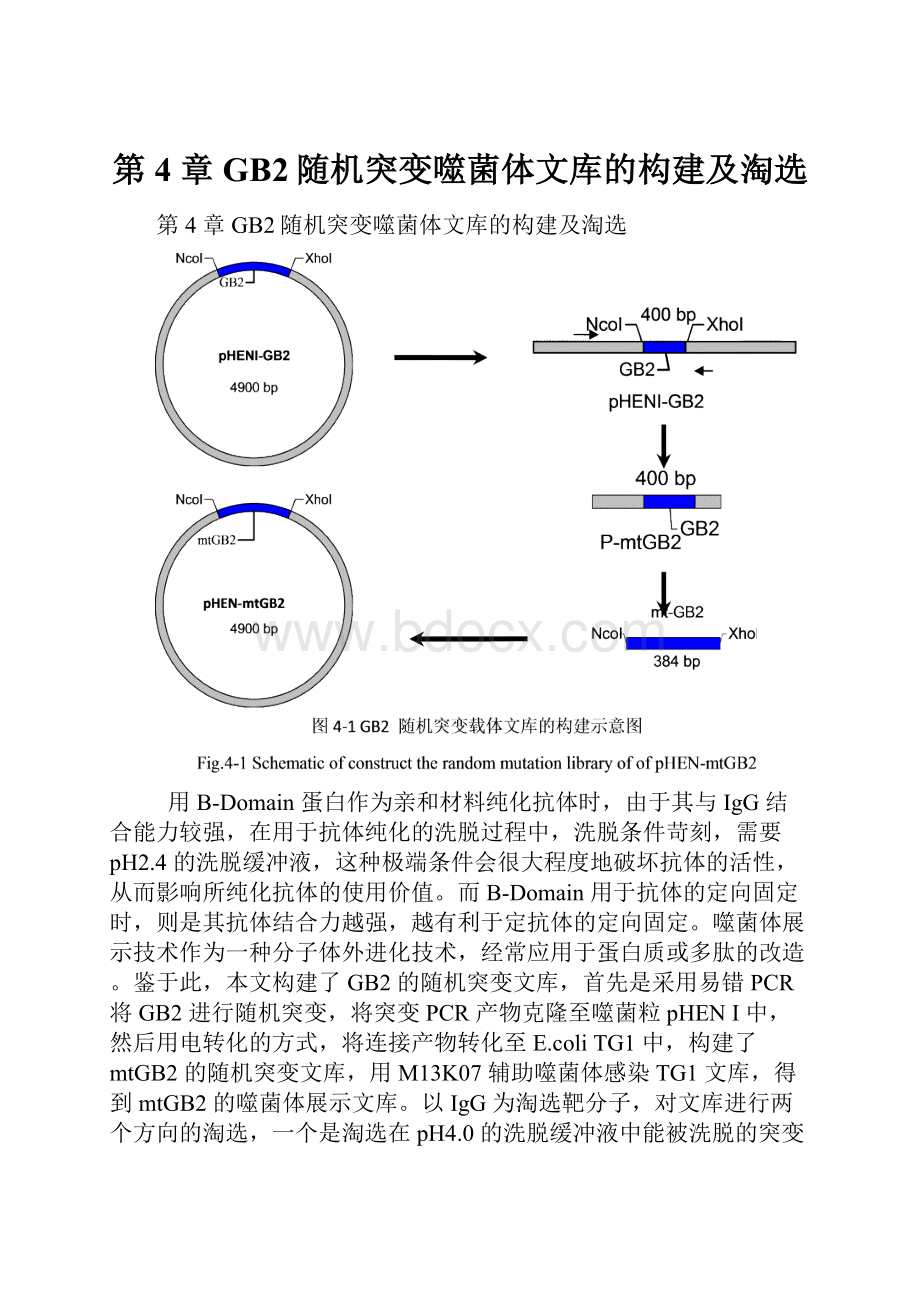
第4章GB2随机突变噬菌体文库的构建及淘选
第4章GB2随机突变噬菌体文库的构建及淘选
用B-Domain蛋白作为亲和材料纯化抗体时,由于其与IgG结合能力较强,在用于抗体纯化的洗脱过程中,洗脱条件苛刻,需要pH2.4的洗脱缓冲液,这种极端条件会很大程度地破坏抗体的活性,从而影响所纯化抗体的使用价值。
而B-Domain用于抗体的定向固定时,则是其抗体结合力越强,越有利于定抗体的定向固定。
噬菌体展示技术作为一种分子体外进化技术,经常应用于蛋白质或多肽的改造。
鉴于此,本文构建了GB2的随机突变文库,首先是采用易错PCR将GB2进行随机突变,将突变PCR产物克隆至噬菌粒pHENI中,然后用电转化的方式,将连接产物转化至E.coliTG1中,构建了mtGB2的随机突变文库,用M13K07辅助噬菌体感染TG1文库,得到mtGB2的噬菌体展示文库。
以IgG为淘选靶分子,对文库进行两个方向的淘选,一个是淘选在pH4.0的洗脱缓冲液中能被洗脱的突变型,能更好地应用于抗体纯化领域;一个是在pH2.4仍不能被洗脱的突变型,能更好地应用于抗体的定向固定。
图4-2亲和力减弱文库A淘选过程示意图
Fig.4-2SchematicofthepanningoflibraryA
图4-3亲和力增强文库B淘选过程示意图
Fig.4-3SchematicofthepanningoflibraryB
4.1材料、仪器与试剂
4.1.1材料
E.coliTG1购自Invitorgen、噬菌粒pHENI、HelpphageM13K07由英国剑桥医学研究委员会(MRC)分子生物学实验室GregWinter博士惠赠;小鼠腹水由本实验室制备。
4.1.2主要仪器
—BIO-RAD凝胶成像系统BIO-RAD公司
—低温离心机(2K-15)Sigma公司
—PCR扩增仪(GeneAmpPCRsystem2400型)PE公司
—型稳压电泳仪(DYY-IⅢ)北京六一仪器厂
—超低温冰箱(CD-196/HC)Thermo公司
—电转仪Bio-Rad公司
—电击杯Bio-Rad公司
—超纯水制备仪(67120)美国Millipore公司
—恒温摇床上海智诚
—96孔酶标板美国Costar公司
—37℃恒温孵育箱日本SANYO
—型全自动灭菌锅(MLS-3750)日本SANYO公司
—酶联免疫检测仪(MK2)Labsystems公司
4.1.3试剂
TaqDNA聚合酶、脱氧核糖核苷酸(dNTPs)、限制性内切酶(XhoⅠ、NcoⅠ、SalⅠ)、DL2000DNAmarker、λ-HindⅢDNAmarker,DNA片段纯化试剂盒、AgaroseGelDNAPurificationKitVer.2.0、质粒提取试剂盒均购买与TaKaRa公司;T4DNAligase购自NEB公司;Goldview核酸染料购自博大泰克生物基因技术有限责任公司;Yeastextract、Tryptone购自Oxoid公司;其他普通化学药品与试剂都是国产分析纯。
4.1.4溶液与培养基
1)LB:
在900mL蒸馏水中加10gTryton,5gYeastExtract,10gNaCl,溶解后加水至一升,用NaOH调节pH至7.4;
2)SOB培养基:
20g/LTrypone,5g/LYeastExtract,0.5g/LNaCl,高压冷却至
55℃后加入1mol/L的无菌MgCl210mL,250mmol/LKCl10mL;
3)SOC培养基:
除含有20mmol/L葡萄糖外,其他成分与SOB培养基相同;
4)SOB平板:
4g胰蛋白胨,1g酵母膏,0.1gNaCl,0.188gKCl,2.03g
MgCl2,0.986gMgSO4,3.6g琼脂粉,加ddH2O至200mL,高压灭菌,冷却后加入200µL100mg/mL氨苄青霉素,混匀后立即铺平皿,4℃保存备用。
5)2×YT:
在900mLddH2O中加16g胰蛋白胨,10g酵母膏,5gNaCl,加ddH2O至1L,调节pH至7.0,高压灭菌备用。
6)2×YT-G:
2×YT培养基中加入2%的葡萄糖;
7)2×YT-AG:
2×Y-GT培养基中加入100µg/mL的氨苄青霉素;
8)2×YT-AK:
2×YT培养基中加入100µg/mL的氨苄青霉素和50µg/mL的卡那霉素;
9)上层琼脂:
每升10g胰蛋白胨,5g酵母膏,5gNaCl,1gMgCl2.6H2O,7g琼脂粉,融化后分装试管3mL/管,高压灭菌,用时用微波炉融化,每加入10µL2M的MgCl2。
10)PEG-NaCl:
20%(w/v)PEG8000,称量200gPEG8000,146.1gNaCl,溶解于800mL去离子水中,定容至1L,高压灭菌,室温储存。
11)PBS:
称取NaCl8.0g,KH2PO40.2g,Na2HPO4·12H2O2.9g,KCl0.2g,加蒸馏水定容至1000mL。
12)洗涤缓冲液PBST:
称取NaCl8.0g,KH2PO40.2g,Na2HPO4·12H2O2.9g,KCl0.2g,加蒸馏水定容至1000mL,再加入Tween-200.5mL。
13)0.1MGly-HCl(pH2.4、4.0、6.0):
称取0.75gGly,溶解于与100mL去离子水中,用HCl溶液分别将pH调节至pH2.4、4.0、6.0。
14)1MTris-HCl(pH9.0)称取12.1gTris,溶解于去离子水中,定容至100mL,调节pH至9.0。
4.2实验方法与步骤
4.2.1野生型pHENI-wtGB2质粒的构建
1)合成引物
pHEN-GB2-NcoI:
GCACCATGGcaACTTACAAATTAATCCTTAATGG(NcoI)
pHEN-GB2-XhoI:
GACCTCGAGTTCAGTTACCGTAAAGGTCTTAGTC(XhoI)
pHEN-F:
CAGGAAACAGCTATGACC
pHEN-R:
GCCCCATTCAGATCCTCTTC
2)以pGEX-GB2为模板,pHEN-GB2-NcoI、pHEN-GB2-XhoI为引物进行PCR,扩增GB2片段。
3)分别NcoI/XhoI双酶切pHENI噬菌粒与PCR产物,分别割胶回收纯化。
4)用T4连接酶将GB2克隆至pHENI噬菌粒中,转化E.coliDH5a细胞。
5)菌落PCR、NcoI/XhoI双酶切验证所得克隆子,选取阳性克隆子送测序。
4.2.2GB2的随机突变
1)易错PCR对mtGB2进行随机突变,进行4组,每组的体系不同,分别降低一种dXTP的浓度至10%,引物为pHEN-F、pHEN-R,每管100μL体系,如下表:
表4-1易错PCR体系
Table4-1ThereactionsystemoferrorPCR
1
2
3
4
其他条件
﹡
﹡
﹡
﹡
dATP
0.02mmol/L
0.2mmol/L
0.2mmol/L
0.2mmol/L
dTTP
0.2mmol/L
0.02mmol/L
0.2mmol/L
0.2mmol/L
dCTP
0.2mmol/L
0.2mmol/L
0.02mmol/L
0.2mmol/L
dGTP
0.2mmol/L
0.2mmol/L
0.2mmol/L
0.02mmol/L
﹡pHENI-wtGB2:
0.2ng;MnCl2:
0.5mmol/L;MgCl2:
5mmol/L;Taq:
5U;上、下引物各20pmol。
反应条件:
94℃变性30s,62℃退火30s,72℃延伸30s,共35循环。
2)将4组易错PCR产物合并,用DNA片段回收试剂盒回收。
3)取部分回收产物进行TA克隆,挑取阳性克隆子送测序,分析随机突变效果。
4.2.3mtGB2与pHENI连接
1)用XhoI和NcoI两种限制性内切酶对mtGB2片段和pHENI噬菌粒进行双酶切,1.0%琼脂糖凝胶电泳后,用割胶试剂盒分别回收纯化,分别定量。
2)连接体系为50µL,按摩尔比5:
1投入GB2与pHENI,2.5µLT4连接Buffer,T4连接酶为0.5µL(350U),16℃反应过夜,共进行4管。
3)向连接产物中加入无水乙醇至终浓度为70%,-20℃沉淀过夜,4℃,10000rpm离心20min,去上清,用70%乙醇洗涤沉淀两次,待乙醇除干净后将沉淀溶解于20µL的无菌水中。
4.2.4电转感受态细胞的制备
1)从LB平板上挑取一个单菌落,接种于5mL的SOB培养基中,37℃振荡培养过夜;
2)按1:
100的比例将TG1过夜培养物接种到500mLSOB培养基中,37℃振荡培养至OD600为0.4;
3)将培养物冰浴30min,4℃,550g离心10min,沉淀细胞;
4)弃上清,用500mL预冷的无菌水水,4℃,550g离心10min,沉淀细胞;
5)弃上清用250mL预冷的无菌10%甘油溶液重悬细胞,4℃,600g离心10min,沉淀细胞;
6)用15mL预冷的无菌10%甘油溶液重悬细胞,4℃、600g离心10min,沉淀细胞;
7)将细胞悬浮在1mL预冷的CYT溶液中,将菌液按50µL/管分装在预冷的0.5mL离心管中,封紧管口,迅速没入液氮中冻存,保存于-70℃。
4.2.5电转化
1)将50µL感受态细胞从冰箱取出,冰上融化,加入2µL连接产物,混匀,冰上放置1min;(将所有的连接产物分多次进行电转);
2)将上述混合液转入0.2cm的电击杯中,调节电击参数:
电压为2.5kv,电场强度:
12.5kv/cm;
3)立即在电击杯中加入1mLSOC培养基,悬浮细胞,37℃培养1h;
4)取5µL适当梯度稀释,每个梯度分别取200µL涂布两块SOB-AG平板,37℃培养过夜,计算两块平板上的菌落数,用于计算库容;
库容=菌落总数/2×稀释倍数×菌液总体积(mL)/0.2×103
5)将剩余的培养物均分涂布平板,使得每块平板上的单菌落的疏密程度合适(不重合,但又不至于太稀),37℃培养过夜;
6)从平板上挑取一些转化子,进行PCR验证,分析插入率。
目的基因插入率=插入GB2的阳性克隆数/挑取的转化子总数×100%
实际库容=库容×目的基因插入率
7)每块平板上加入1mL2×YT培养基,用灭菌的药匙将细菌洗脱,将所有平板的细菌悬液合并,混匀。
取5mL细菌悬液用于噬菌体挽救,剩余的菌液则制备成20%的甘油保存液,–70℃冻存,此即为GB2随机突变大肠杆菌文库。
4.2.6辅助噬菌体滴度的测定
1)接种TG1于5mL2×YT培养基中,摇床培养至对数期后置于4℃预冷。
(可保存一周)
2)将试管中的3mL上层琼脂融化,将其温度平衡至48℃,每测一个滴度准备一管。
3)用2×YT培养基对待测辅助噬菌体噬菌体进行10倍梯度稀释。
4)将预冷的TG1培养物分装于1.5mL离心管中,每管100µL,每个稀释梯度一管。
5)将100µL不同稀释度的噬菌体分别加入到TG1培养物中,快速振荡混匀,37℃温育5min。
6)将温育后的混合物加入至上层琼脂中,迅速混匀后倾倒在2×YT平板上,待培养基凝结后倒置平板,37℃培养过夜,计算平板上的噬菌斑数量,以噬菌斑为30-300个左右的平板计数为准,计算噬菌体溶液的滴度。
4.2.7M13K07辅助噬菌体的扩增
1)从噬菌体滴度测定平板上,用牙签挑取一个独立的、噬菌斑较大的噬菌斑接种到5mL2×YT-K试管中,37℃、250rpm培养12h。
2)取500µL培养物接种到装有50mL2×YT-K培养基的三角瓶中,30℃、250rpm培养过夜。
3)收集过夜培养物,10000rpm离心20min,收集上清,含有噬菌体。
4)加入1/5体积的PEG/NaCl,充分混匀,让噬菌体4℃沉淀至少60min,最好过夜;
5)10000rpm离心20min,去上清,收集沉淀,溶解于8mL无菌PBS中。
6)测定M13K07溶液的滴度。
4.2.8GB2随机突变噬菌体文库的构建
1)将5mL细菌悬液转移至一50mL的离心管中,用2×YT培养基调整OD600至0.3,记录最终体积。
加入Amp使其终浓度为100µg/mL,加入无菌葡萄糖使其终浓度为2%,37℃振荡培养1h;
2)按phage:
cell=20:
1加入辅助噬菌体,37℃振荡培养2h;
3)1000g离心10min,弃上清,用50mL2×YT-AK培养基轻轻悬浮细胞,30℃振荡培养过夜;
4)收集培养物,10000g离心20min,将上清转移至10mL离心管中,上清中含有重组噬菌体;
5)加入1/5体积的PEG/NaCl,充分混匀,让噬菌体4℃沉淀至少60min,最好过夜;
6)4℃、1000g离心20min,倒掉上清,再次短暂离心,吸去残留的上清;
7)加入2mLPBS-1%BSA溶液重悬细胞,悬液转入10mL离心管中,4℃离心5min,沉淀残余的细胞;吸取上清,为并分装于1.5mL离心管中,加入50%甘油,-70℃保存备用。
此即为原始的GB2随机突变噬菌体文库;
8)测定文库滴度。
4.2.9噬菌体展示文库滴度的测定及无菌实验
1)接种TG1于5mL2×YT培养基中,摇床培养至对数期;
2)用SOB培养基10倍梯度稀释待测噬菌体文库;
3)待TG1培养物达到对数中期时,将其200µL等分于1.5mL离心管中,每个稀释梯度一管;
4)每管加入10µL的不同稀释度的噬菌体,快速振荡混匀,37℃温育10min,
5)将孵育后产物涂布于SOB-AG平板上,37℃倒置培养过夜。
6)计算平板上的单菌落数,计算噬菌体文库的滴度;
7)取10µL噬菌体涂布平板,37℃培养过夜,看是否有菌落生长。
4.2.10筛选靶分子鼠IgG的纯化
1)收集1mL小鼠腹水,4℃10000rpm离心10min,除去沉淀,将上清用0.45µm滤膜过滤,除去不溶性的小颗粒。
2)取1.5mLProteinGSepharose加入到层析柱中,用10倍体积的PBS进行过柱洗涤,注意液面,加上塞子,防止树脂暴露于空气中干燥。
3)加入0.5mL过滤后的小鼠腹水与0.5mLPBS混匀,盖上盖、塞后室温轻轻混匀15min,然后除去盖、塞,让液体流出。
4)加入10倍体积的PBS洗涤层析柱中树脂。
5)加入1mL0.1MGly-HCl(pH2.5)洗脱缓冲液,过柱洗脱,收集洗脱液,迅速加入适量的1MTris-HCl(pH9.0)中和缓冲液进行中和。
6)将获得的IgG洗脱液进行SDS-PAGE分析其纯度和浓度。
4.2.11IgG结合力减弱文库A的淘选
1)用PBS稀释IgG至终浓度为5μg/mL,按100µL/孔加入酶标孔中,4℃包被过夜。
2)PBST洗涤3次,加入2%BSA37℃封闭2h。
3)PBST洗涤3次,加入100µLPHEN-mtGB2噬菌体展示文库,噬菌体数量为1011cfu,37℃孵育1.5h。
4)用pH6.0的PBS洗涤缓冲液洗涤3次,然后加入300µL该缓冲液,37℃12min。
5)用pH6.0的PBS洗涤缓冲液洗涤10次,加入200µLpH4.0的Gly-HCl洗脱缓冲液,37℃孵育25min,将该洗脱缓冲液转移至一无菌离心管中,用中和缓冲液中和。
6)取10µL进行梯度稀释,测定滴度,计算淘选回收率,其余则加入至1mLTG1对数期培养物进行感染,37℃孵育1h,保存备用进行文库的扩增。
7)将文库扩增结果进行下一轮淘选,改变淘选条件,每一轮淘选条件如表4-2。
表4-2亲和力减弱文库A(pH4.0-6.0)各轮的淘选条件
Tab.4-2ThepanningconditionofeachroundsofthelibraryA(pH4.0-6.0)
轮次
IgG包被量(µg)
文库投入量(cfu)
洗涤时间(min)
洗脱时间(min)
1
0.5
1.0×1011
12
25
2
0.25
1.0×1011
15
25
3
0.1
2.0×1010
20
20
4
0.1
1.0×1010
20
15
4.2.12IgG结合增强文库B的淘选
1)用PBS稀释IgG至终浓度为5µg/mL,按100µL/孔加入酶标孔中,4℃包被过夜。
2)PBST洗涤3次,加入2%BSA37℃封闭2h。
3)PBST洗涤3次,加入100µLpHEN-mtGB2噬菌体展示文库,噬菌体数量为1011cfu,37℃孵育1.5h。
4)用pH7.0的PBST洗涤缓冲液洗涤10次。
5)用0.1MGly-HCl(pH2.4)洗涤缓冲液洗涤3次,加入300µL该缓冲液,37℃孵育12min,再用0.1MGly-HCl(pH2.4)洗涤缓冲液洗涤3次,然后用pH7.0的PBST洗涤缓冲液洗涤3次。
6)加入200µLTG1对数期培养物,37℃感染1h。
7)取出感染后TG1培养物,取10µL进行梯度稀释,测定滴度,计算淘选回收率,其余则进行文库的扩增。
8)将扩增后文库进行下一轮淘选,改变淘选条件,每一轮淘选条件如表4-3。
表4-3亲和力减弱文库B(pH<pH2.4)各轮的淘选条件
Tab.4-3ThepanningconditionofeachroundsofthelibraryB(pH<pH2.4)
轮次
IgG包被量(µg)
文库投入量(cfu)
洗涤时间(min)
1
0.5
1.0×1011
12
2
0.25
2.0×1010
15
3
0.1
5.0×109
20
4
0.1
5.0×109
25
4.2.13淘选后文库的扩增
1)将文库感染物产物加入至3mL2×YT-G培养基中,37℃、225rpm振荡培养1h,加入Amp至终浓度为100ng/mL,同时按cell:
phage=1:
20的比例加入M13K07噬菌体,37℃、225rpm振荡培养2h。
2)将培养物分装于离心管中,1000rpm,5min,收集菌体,加入至20mL2×YT-AK培养液中,30℃,250rpm振荡培养过夜。
3)将过夜培养物10000rpm离心20min,收集上清,每5mL上清中加入1mLPEG/NaCl,混匀后置于4℃1h以上,最好是过夜。
4)10000rpm,20min,去除上清,将沉淀重悬于2mLTBS中,加入1/5体积的PEG/NaCl,混匀后置于4℃1h以上。
5)10000rpm,20min,去除上清,将沉淀悬浮于400µLTBS-1%BSA中,即为该轮淘选后文库,测定滴度,用于下一轮淘选或者分析。
4.3实验结果
4.3.1野生型pHENI-GB2质粒的构建
通过NcoI和XhoI将野生型wtGB2克隆至pHENI中,菌落PCR验证后提取阳性克隆子的质粒,用NcoI和XhoI进行双酶切验证,结果获得两条带,分别与空载体和外源片段大小一致,说明载体构建成功(图4-4)。
进一步经测序确正,未发现突变。
图4-4pHENI-wtGB2重组质粒的NcoI和XhoI双酶切验证图
Fig.4-4IdentificationofthepHENI-wtGB2byrestrictionenzymeofNcoIandXhoI
M1:
λ-HindⅢdigestDNAmarker;M2:
DL2000marker;
1:
pHENI噬菌粒双酶切样品;2:
pHENI-wtGB2重组噬菌粒双酶切样品;
3:
mtGB2DNA片段双酶切样品
4.3.2GB2的随机突变
以pHENI-wtGB2重组噬菌粒为模板,pHEN-F和pHEN-R为引物,采用易错PCR方法对GB2进行随机突变,分四管进行,四管分别将一种脱氧核苷酸的浓度降低90%,每个反应都进行35个循环。
1.0%琼脂糖凝胶电泳分析,四个反应均成功进行,产物浓度、大小也一致(见图4-5)。
图4-5易错PCR扩增产物的琼脂糖凝胶电泳图
Fig.4-5Analysisoftheerror-PCRproducts
M:
DL2000Marker;1、2、3、4:
分别为减少90%的dATP、dTTP、dCTP和dGTP的易错PCR扩增产物;
将4管易错PCR产物混合,经乙醇沉淀浓缩纯化,取部分进行TA克隆,经菌落PCR验证后,挑取4个克隆子送测序,用DNASTAR软件将其与野生型GB2片段进行比较,分析随机突变情况。
由图4-6可以看出共发生了29处点突变,突变率约为1.93%。
随机突变位点的分布位置是随机的,没有出现突变热点。
就突变碱基而言,主要是A、T发生突变,占了约82%,突变结果表现出了一定的偏向性。
图4-6随机突变产物的氨基酸突变结果分析图
Fig.4-6ComparisonoftheaminoacidsequencesbetweenwtGB2andfourmtGB2clonesfromerror-PCR
黄色:
发生点突变;红色:
未发生点突变