基因芯片检测技术Word文档格式.docx
《基因芯片检测技术Word文档格式.docx》由会员分享,可在线阅读,更多相关《基因芯片检测技术Word文档格式.docx(10页珍藏版)》请在冰豆网上搜索。
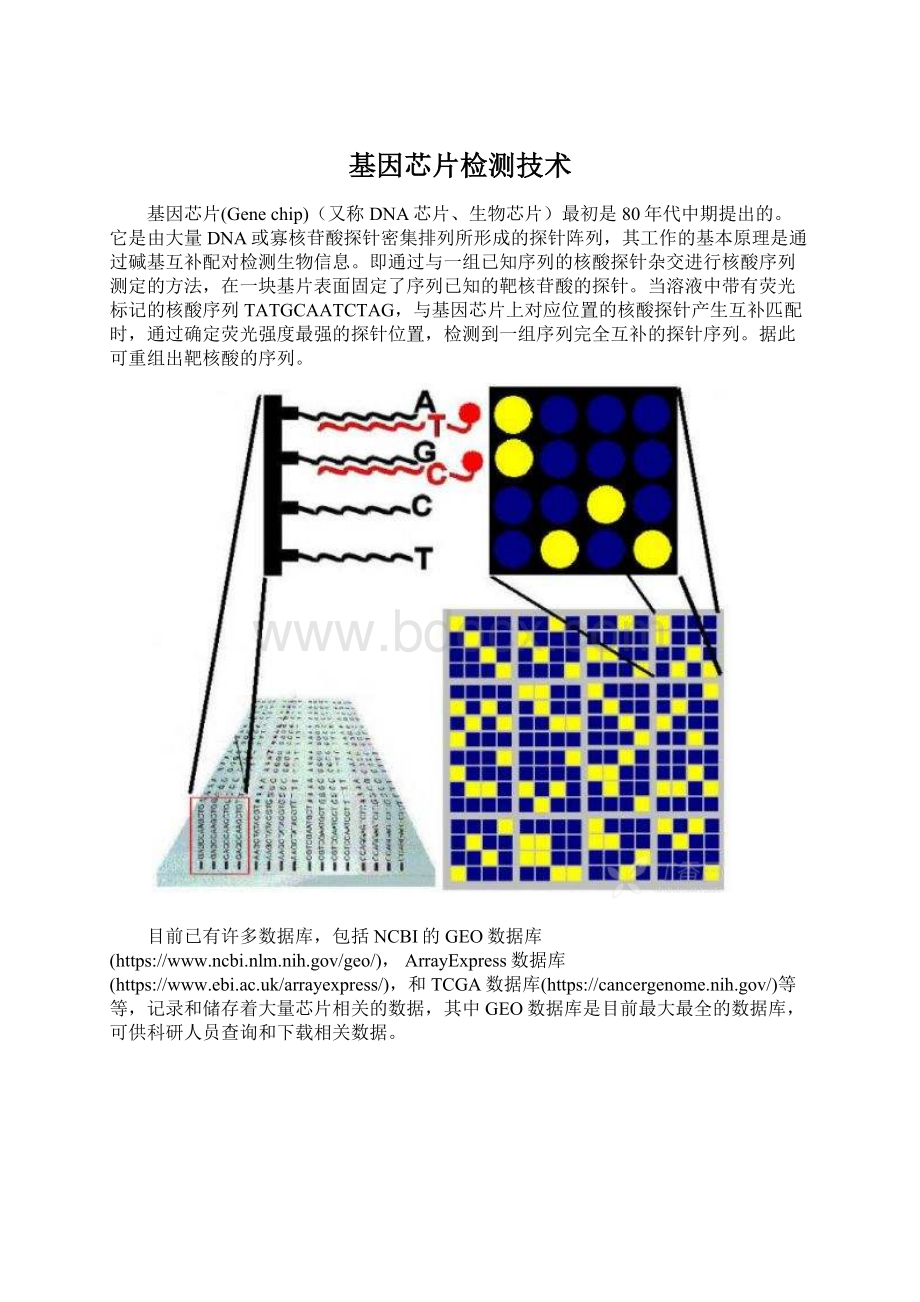
背景处理之后,我们可以将芯片数据放入一个矩阵中:
其中,各字母的意义如下:
N:
条件数;
G:
基因数目(一般情况下,G>
>
N);
行向量mi=(mi1,mi2,…,miN)表示基因i在N个条件下的表达水平(这里指绝对表达水平,亦即荧光强度值);
列向量mj=(m1j,m2j,…,mGj)表示在第j个条件下各基因的表达水平(即一张芯片的数据);
元素mij表示第基因i在第j个条件下(绝对)基因表达数据。
m可以是R(红色,Cy5,代表样品组)。
也可以是G(绿色,Cy3,代表对照组)。
2)芯片数据清理:
经过背景校正后的芯片数据中可能会产生负值,还有一些单个异常大(或小)的峰(谷)信号(随机噪声)。
对于负值和噪声信号,通常的处理方法就是将其去除,常见数据经验型舍弃方法有:
A.标准值或奇异值舍弃法;
B.变异系数法;
前景值<200;
前景值-平均数/前景值-中位数<80%等等。
然而,数据的缺失对后续的统计分析(尤其是层式聚类和主成分分析)有致命的影响。
Affymetrix公司的芯片分析系统会直接将负值修正为一个固定值。
缺失值得处理方法:
对数据的删除,通常是删去所在的列向量或行向量。
一个比较常用的做法是,事先定义个阈值M。
若行(列)向量中的缺失数据量达到阈值M,则删去该向量。
若未达到M,有两种方法处理,一是以0或者用基因表达谱中的平均值或中值代替,另一个是分析基因表达谱的模式,从中得到相邻数据点之间的关系,据此利用相邻数据点估算得到缺失值(类似于插值)。
填补缺失值(k临近法):
利用与待补缺基因距离最近的k个临近基因的表达值来预测待填补基因的表达值。
3)提取芯片数据的表达值:
由于芯片数据的小样本和大变量的特点,导致数据分布呈偏态、标准差大。
对数转换能使上调、下调的基因连续分布在0的周围,更加符合正态分布,同时对数转换使荧光信号强度的标准差减少,利于进一步的数据分析。
4)芯片数据的归一化:
经过背景处理和数据清洗处理后的修正值反映了基因表达的水平。
然而在芯片试验中,各个芯片的绝对光密度值是不一样的,在比较各个试验结果之前必需将其归一化(normalization,也称作标准化)。
数据的归一化目的是调整由于基因芯片技术引起的误差,不是调整生物RNA样本的差异。
在同一块芯片上杂交的、由不同荧光分子标记的两个样品间的数据,也需归一化。
常用的方法是平均数、中位数标准化(meanormediannormalization):
将各组实验的数据的logratio中位数或平均数调整在同一水平。
中位数标准化:
将每个芯片上的数值减去各自芯片上logRatio值的中位数,使得所有芯片的logRatio值中位数就变成了0,从而不同芯片间logRaito具有可比性。
5)差异基因表达分析:
经过预处理,探针水平数据转变为基因表达数据。
为了便于应用一些统计和数学术语,基因表达数据仍采用矩阵形式。
A.芯片数据的差异分析主要包括三种方法:
1.倍数分析方法:
倍数变换foldchange,单纯的case与control组表达值相比较,对没有重复实验样本的芯片数据,或者双通道数据采用这种方法。
2.参数法分析(t检验):
当t超过根据可信度选择的标准时,比较的两样本被认为存在着差异。
但小样本基因芯片实验会导致不可信的变异估计,此时采用调节性T检验。
3.非参数分析:
由于微阵列数据存在“噪声”干扰而且不满足正态分布假设,用t检验有风险。
非参数检验并不要求数据满足特殊分布的假设,所以可使用非参数方法对变量进行筛选。
如经验贝叶斯法、芯片显著性分析SAM法。
B.芯片数据的差异分析的常用软件包括:
1.Limma:
它是一个功能比较全的包,既含有cDNA芯片的RAWdata输入、前处理(归一化)功能,同时也有差异化基因分析的“线性”算法(limma:
LinearModelsforMicroarrayData),特别是对于“多因素实验(multifactordesignedexperiment)”。
limma包的可扩展性非常强,单通道(onechannel)或者双通道(towchannel)数据都可以分析差异基因,甚至也包括了定量PCR和RNA-seq。
2.DESeq2和EdgeR包:
都可用于做基因差异表达分析,主要也是用于RNA-Seq数据,同样也可以处理类似的ChIP-Seq,shRNA以及质谱数据。
这两个都属于R包,其相同点在于都是对countdata数据进行处理,都是基于负二项分布模型。
上述两个包通常通过R语言软件实现分析,在R以及bioconductor中,都有对应的分析包使用。
3.GFOLD软件:
对于有生物学重复的数据(一般的转录组数据都会有生物学重复),我们一般采用一个叫edgeR和DEseq的R包。
但如果预先测了一批数据没有重复的数据进行一个预分析。
这时候edgeR依然可以用,不过需要认为指定一个dispersion值,这样的不同的人就可以有不同的结果,在查阅了很多资料之后呢,大家一致认为没有重复的转录组数据应该用GFOLD软件。
C.差异分析后数据的分析
一般获取差异基因后,会对获得的基因进行功能分析,目前常用的功能分析方法和工具包括以下几种:
一、GO基因本体论分类法
最先出现的芯片数据基因功能分析法是GO分类法。
GeneOntology(GO,即基因本体论)数据库是一个较大的公开的生物分类学网络资源的一部分,它包含38675
个EntrezGene注释基因中的17348个,并把它们的功能分为三类:
分子功能,生物学过程和细胞组分。
在每一个分类中,都提供一个描述功能信息的分级结构。
这样,GO中每一个分类术语都以一种被称为定向非循环图表(DAGs)的结构组织起来。
研究者可以通过GO分类号和各种GO数据库相关分析工具将分类与具体
基因联系起来,从而对这个基因的功能进行描述。
在芯片的数据分析中,研究者可以找出哪些变化基因属于一个共同的GO功能分支,并用统计学方法检定结果是否
具有统计学意义,从而得出变化基因主要参与了哪些生物功能。
EASE(ExpressingAnalysisSystematicExplorer)是比较早的用于芯片功能分析的网络平台。
由美国国立卫生研究院(NIH)的研究人员开发。
研究者可以用多种不同的格式将芯片中得到的基因导入EASE
进行分析,EASE会找出这一系列的基因都存在于哪些GO分类中。
其最主要特点是提供了一些统计学选项以判断得到的GO分类是否符合统计学标准。
EASE
能进行的统计学检验主要包括Fisher
精确概率检验,或是对Fisher精确概率检验进行了修饰的EASE
得分(EASEscore)。
由于进行统计学检验的GO分类的数量很多,所以EASE采取了一系列方法对“多重检验”的结果进行校正。
这些方法包括弗朗尼校正法(Bonferroni),本杰明假阳性率法(Benjaminifalsediscoveryrate)和靴带法(bootstraping)。
同年出现的基于GO分类的芯片基因功能分析平台还有底特律韦恩大学开发的Onto-Express。
2002年,挪威大学和乌普萨拉大学联合推出的Rosetta系统将GO分类与基因表达数据相联系,引入了“最小决定法则”(minimaldecisionrules)的概念。
它的基本思想是在对多张芯片结果进行聚类分析之后,与表达模式不相近的基因相比,相近的基因更有可能参与相同的生物学功能的实现。
GCBI分析平台:
是一个新型的网络分析平台,全称是Gene-CloudofBiotechnologyInformation。
GO-Analysis是对基因进行显著性功能(GO)的分析。
由于GO的条目中包含功能的层级关系,从而GO中包含基因的数目变化较大,通常在一到几百个基因之间。
GCBI的功能分析主要用Fisher精确检验,即利用如下的四格表:
在这个假设下分别利用fisher精确检验和检验,分别得到值和值,通过多重比较检验,确定GO的FDR。
最后得出显著性GO,完成GO-Analysis。
ENRICHMENT计算公式为:
比较著名的基于GO分类法的芯片数据分析网络平台还有七十多个,表1列举了其中的一部分。
Name
InternetSite
GCBI
Onto-Tools
http:
//vortex.cs.wayne.edu/projects.htm
ROSETTA
//rosetta.lcb.uu.se/general/
GOToolBox
//burgundy.cmmt.ubc.ca/GOToolBox/
GOstat
//gostat.wehi.edu.au/
GFINDer
//www.medinfopoli.polimi.it/GFINDer/
FatiGO
//www.fatigo.org/
EASE
//david.abcc.ncifcrf.gov/ease/ease.jsp
表1
用GO
分类法进行芯片功能分析的网络平台
二、pathway通路分析法
通路分析是现在经常被使用的芯片数据基因功能分析法。
与GO分类法(应用单个基因的GO分类信息)不同,通路分析法利用的资源是许
多已经研究清楚的基因之间的相互作用,即生物学通路。
研究者可以把表达发生变化的基因列表导入通路分析软件中,进而得到变化的基因都存在于哪些已知通路中,并通过统计学方法计算哪些通路与基因表达的变化最为相关。
现在已经有丰富的数据库资源帮助研究人员了解及检索生物学通路,对芯片的结果进行分析。
主要的生物学通路数据库有以下两个:
①KEGG 数据库:
迄今为止,KEGG数据库(Kyotoencyclopediaofgenesandgenomes)是向公众开放的最为著名的生物学通路方面的资源网站。
在这个网站中,每一种生物学通路都有专门的图示说明。
②BioCarta
数据库:
BioCarta
是一家生物技术公司,它在其公共网站上提供了用于绘制生物学通路的模板。
研究者可以把符合标准的生物学通路提供给BioCarta数据库。