绿色荧光蛋白GFP基因的克隆和表达新手详细注释版.docx
《绿色荧光蛋白GFP基因的克隆和表达新手详细注释版.docx》由会员分享,可在线阅读,更多相关《绿色荧光蛋白GFP基因的克隆和表达新手详细注释版.docx(21页珍藏版)》请在冰豆网上搜索。
绿色荧光蛋白GFP基因的克隆和表达新手详细注释版
绿色荧光蛋白(GFP)基因的克隆和表达
背景知识
绿色荧光蛋白(greenfluorescentprotein,GFP)是一类存在于包括水母、水螅和珊瑚等腔肠动物体内的生物发光蛋白。
当受到紫外或蓝光激发时,GFP发射绿色荧光。
它产生荧光无需底物或辅因子。
发色团是其蛋白质一级序列固有的。
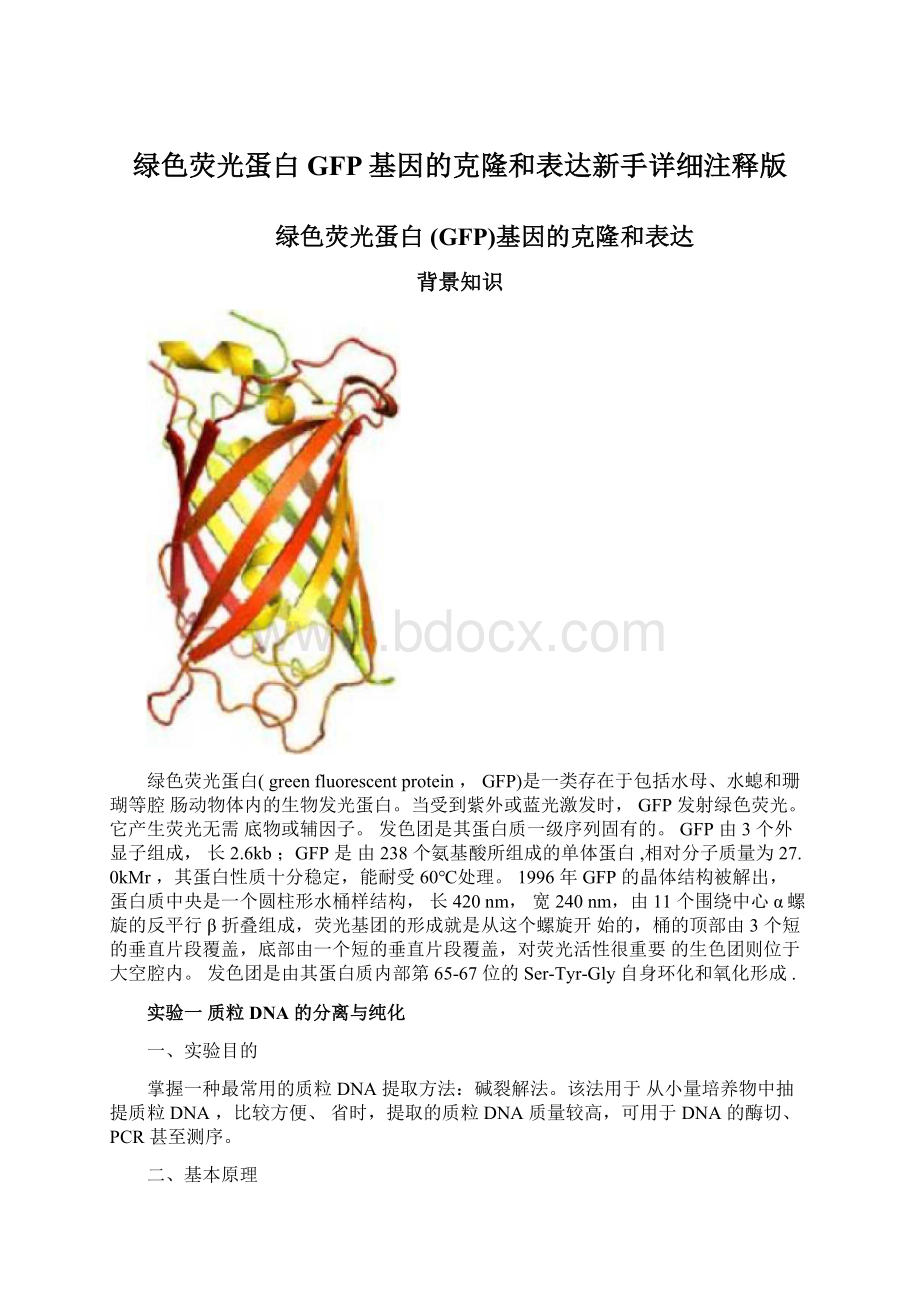
GFP由3个外显子组成,长2.6kb;GFP是由238个氨基酸所组成的单体蛋白,相对分子质量为27.0kMr,其蛋白性质十分稳定,能耐受60℃处理。
1996年GFP的晶体结构被解出,蛋白质中央是一个圆柱形水桶样结构,长420nm,宽240nm,由11个围绕中心α螺旋的反平行β折叠组成,荧光基团的形成就是从这个螺旋开始的,桶的顶部由3个短的垂直片段覆盖,底部由一个短的垂直片段覆盖,对荧光活性很重要的生色团则位于大空腔内。
发色团是由其蛋白质内部第65-67位的Ser-Tyr-Gly自身环化和氧化形成.
实验一质粒DNA的分离与纯化
一、实验目的
掌握一种最常用的质粒DNA提取方法:
碱裂解法。
该法用于从小量培养物中抽提质粒DNA,比较方便、省时,提取的质粒DNA质量较高,可用于DNA的酶切、PCR甚至测序。
二、基本原理
10-200个拷贝。
当宿主细胞的蛋白
质粒是一类在细菌细胞内发现的独立于染色体外,能够自主复制的稳定的遗传单位。
迄今为止,从细菌中分离得到的质粒都是环型双链DNA分子,分子量范围从1kb到200kb。
质粒DNA可持续稳定地处于染色体外的游离状态,但在一定条件下又会可逆地整合到寄主染色体上,随着染色体的复制而复制,并通过细胞分裂传递到后代。
在大多数情况下质粒DNA复制中的酶体系和细菌染色体复制时所用的酶是相同的。
有些质粒复制受宿主细胞复制作用的严格限制,因此每个细胞中只含一个或几个拷贝,称为严谨型质粒,有的质粒的复制受宿主细胞的控制不严,称为松弛型质粒,它们在每个细胞中的数目可达质合成受到抑制时(例如经氯霉素处理),细菌染色体虽不再增加,但松弛型质粒DNA可继续被复制,以至每个细胞内的拷贝数可以增至一千到几千。
质粒具有一定的生物功能,它们往往带有一些抗药标记,当质粒DNA用人为的方法转化进细菌时,转化后的细菌会表现出质粒基因所具有的新的生物表现型,例如,把一个含有抗药基因的质粒转入细菌后,原来无抗药性的细菌则表现出抗药的新表型。
借助转化菌获得的新表型特征,可证实质粒已转入宿主细菌中,这样就可以作为转化菌的选择性标记。
质粒作为基因克隆载体分子的重要的条件是获得批量的纯化的质粒DNA分子。
目前已有许多方法可用于质粒DNA的提取,它们都包括三个基本的步骤:
细菌的生长和质粒的扩增;菌体的收集裂解,质粒DNA的分离;质粒DNA的纯化。
1、细菌的生长和质粒的扩增从琼脂培养基平板上挑取一个单菌落,接种到含适当抗生素的液体培养基中培养。
对于松弛型质粒(如pUC系列)来说,只要将培养物放到标准的LB或2YT培养基中生长到对数晚期,就可以大量提取质粒,而不必选择性地扩增质粒DNA。
但对于严谨型质粒(如pBR322)来说,则需在得到部分生长的细菌培养物中加入氯霉素继续培养若干小时,以便对质粒进行选择性扩增。
2、菌体的收集、裂解和质粒DNA的分离质粒分离的基本原理是利用宿主菌(一般是大肠杆菌菌株)DNA与质粒DNA之间的两种主要性质差异:
(1)大肠杆菌的染色体较一般的载体质粒DNA大得多。
(2)从细胞中提取得
到的大肠杆菌DNA主体是变性的线性分子,而大多数质粒DNA是共价闭合的环状分子。
这里主要介绍碱裂解法的基本原理:
在细菌悬浮液中加入SDS(十二烷基硫酸钠)和NaOH使菌体
裂解(有时需要先使用溶菌酶水解细胞壁)。
此处理可破坏碱基配对,故可使细菌的线状染色体DNA变性,但闭环质粒DNA链由于处于拓扑缠绕状态而不能彼此分开。
当条件恢复正常时(如加入酸性的NaAc或Kac中和碱性NaOH),质粒DNA链迅速得到准确配对,重新恢复成天然的超螺旋分子。
通过离心,可以使染色体DNA与变性蛋白质、RNA分子一起沉淀下来,而质粒超螺旋分子仍滞留于上清中。
3、质粒DNA的提纯
胶电泳中不同构型的同一种质粒DNA,尽管分子量相同,但具有不同的电泳迁移率。
其中走在
最前沿的是SCDNA,其后依次是LDNA和OCDNA。
三、实验材料、仪器及试剂
1.在含有pEGFP-N3质粒的DH5α平板上菌落上挑取菌种,置于含有5mLLB培养基的试管中。
摇晃过夜。
(DH5α是一种大肠杆菌的诱变菌株,主要表现对外源DNA的免疫缺乏,是用
于基因工程的菌种)
在含有pET-28a质粒的平板上挑取单菌落于另外一个试管中,同样摇荡培养过夜。
2、使用仪器恒温培养箱,超净台,恒温摇床,制冰机,台式离心机,小型混合器,冰箱
四、实验步骤
1.取1.5mlDH5α培养液倒入1.5mLeppendorf管(一种离心管)中,13000rpm离心1min。
2.重复1。
3.弃上清,将管倒置于卫生纸上数分钟,使液体流尽。
4.菌体沉淀重悬浮于100μL溶液Ⅰ中(需剧烈振荡),室温下放置10min。
(溶液I,50mM葡萄糖/25mMTris-Cl/10mMEDTA,pH8.0;溶液I的作用;mM为mmol/L。
任何生物化学反应,首先要控制好溶液的pH,因此用适当浓度的和适当pH值的Tris-Cl溶液。
50
mM葡萄糖最大的好处是悬浮后的大肠杆菌不会快速沉积到管子的底部。
EDTA是Ca2+和Mg2+等二
价金属离子的螯合剂,配在分子生物学试剂中的主要作用是:
抑制DNase的活性,和抑制微生物
生长。
在溶液I中加入高达10mM的EDTA,无非就是要把大肠杆菌细胞中的所有二价金属离子都螯合掉。
菌体一定要悬浮均匀,不能有结块。
)
5.加入新配制的溶液Ⅱ200μl,盖紧管口,快速温和颠倒eppendorf管5次,以混匀内容物(千万不要振荡),冰浴5min。
(溶液II,0.2MNaOH/1%SDS;溶液II的作用;这是用新鲜的0.4N的NaOH和2%的SDS等体积混合后使用的。
要新从浓NaOH稀释制备0.4N的NaOH,保证NaOH没有吸收空气中的CO2而减弱碱性。
其实破细胞的主要是碱,而不是SDS,所以才叫碱法抽提。
事实上NaOH是最佳的溶解细胞的试剂,不管是大肠杆菌还是哺乳动物细胞,碰到了碱都会几乎在瞬间就溶解,这是由于细胞膜发生了从bilayer(双层膜)结构向micelle(微囊)结构的相变化所导致。
用了不新鲜的0.4NNaOH,即便是有SDS也无法有效溶解大肠杆菌(不妨可以自己试一下),自然就难高效率抽提得到质粒。
这一步要记住两点:
第一,时间不能过长,因为碱性条件下基因组DNA片断会慢慢断裂;第二,必须温柔混合,不然基因组DNA也会断裂。
)
6.加入150μL预冷的溶液Ⅲ,盖紧管口,并倒置离心管,温和振荡3次,使沉淀混匀,冰浴中15分
钟,13000rpm离心5min。
(溶液III,3M醋酸钾/2M醋酸。
溶液Ⅲ含3M醋酸钾/2M醋酸。
溶液III加入后就会有大量的沉淀,这其实是SDS遇到钾离子后变成了十二烷基硫酸钾(PDS),而PDS是水不溶的,因
此发生了沉淀。
而高浓度的盐,使得沉淀更完全。
SDS易与蛋白质结合,平均两个氨基酸上结合
一个SDS分子,钾钠离子置换所产生的大量沉淀自然就将绝大部分蛋白质沉淀。
大肠杆菌的基因组DNA也会一起被共沉淀,因为基因组DNA太长,容易被PDS共沉淀,注意SDS并不与DNA分子结合。
2M的醋酸是为了中和NaOH,因为长时间的碱性条件会打断DNA,所以要中和之。
基因组DNA一旦
发生断裂,只要是50-100kb大小的片断,就没有办法再被PDS共沉淀了。
所以碱处理的时间要短,而且不得激烈振荡,不然最后得到的质粒上总会有大量的基因组DNA混入,琼脂糖电泳可以
观察到一条浓浓的总DNA条带。
很多人误认为是溶液III加入后基因组DNA无法快速复性就被沉淀了,这是天大的误会,因为变性的也好复性的也好,DNA分子在中性溶液中都是溶解的。
NaOH
本来是为了溶解细胞而用的,DNA分子的变性其实是个副产物,与它是不是沉淀下来其实没有关系。
溶液III加入并混合均匀后在冰上放置,目的是为了PDS沉淀更充分一点。
)
7.上清液移入干净eppendorf管中,加入等体积的氯仿/异戊醇(24:
1),振荡混匀,13000rpm离心2min。
(PDS沉淀的形成后有些蛋白质不能被沉淀,因此要用酚/氯仿/异戊醇进行抽提,然后进行酒精沉淀才能得到质量稳定的质粒DNA,不然时间一长就会因为混入的DNase而发生降解。
这里用25:
24:
1的酚/氯仿/异戊醇是有很多道理的。
酚(Phenol)对蛋白质的变性作用远大于氯仿,但是水饱和酚的比重略比水重,碰到高浓度的盐溶液(比如4M的异硫氰酸胍),离心后酚相会跑到
上层,不利于含质粒的水相的回收;加入氯仿后可以增加比重,使得酚/氯仿始终在下层,方便水相的回收;还有一点,酚与水有很大的互溶性,如果单独用酚抽提后会有大量的酚溶解到水相中,而酚会抑制很多酶反应(比如限制性酶切反应),而用酚/氯仿的混合液进行抽提,跑到水相中的酚则少得多,微量的酚在乙醇沉淀时就会被除干净而不必担心酶切等反应不能正常进行。
至于异戊醇的添加,其作用主要是为了让离心后上下层的界面更加清晰,也方便了水相的回收。
)
8.将水相移入干净eppendorf管中,加入等体积的氯仿/异戊醇(24:
1)振荡混匀,13000rpm离心2min。
9.将水相移入干净eppendorf管中,加入2倍体积的无水乙醇,振荡混匀后,置于室温下2min,然后13000rpm离心5min。
(回收后的水相含有足够多的盐,因此只要加入2倍体积的乙醇,在室温放置几分钟后离心就可以将质粒DNA沉淀出来。
)
10.弃上清,将管口敞开倒置于卫生纸上使所有液体流出,加入1mL70%乙醇洗沉淀一次,振荡混
匀后,13000rpm离心5min。
(高浓度的盐会水合大量的水分子,因此DNA分子之间就容易形成氢键而发生沉淀。
如果感觉发
生了盐的沉淀,就用70%的乙醇多洗几次,每次在室温放置一个小时以上,并用移液枪枪头(tip)将沉淀打碎,就能得到好的样品。
)
11.吸除上清液,将管倒置于卫生纸上使液体流尽,室温干燥。
12.将沉淀溶于30μLTE缓冲液(pH8.0,含20μg/mLRNaseA)中,保存在-20℃冰箱中。
(溶解已经讲解的RNA,防止未降解的RNA会干扰电泳结果。
)
13.按照同样的流程和方法将pET-28a的质粒也提出来,保存在-20℃冰箱中。
五、实验结果
实验二质粒DNA浓度的测定
、实验目的
学习利用核酸蛋白测定仪测算核酸的浓度和纯度。
、基本原理
核酸分子在260nm下有最大吸光值,因此可以通过260nm下核酸的吸光值计算核酸浓度
mg/ml),并通过测定与280nm和230nm的比值,估算DNA的纯度。
除了核酸浓度,分光光度计同时显示几个非常重要的比值表示样品的纯度,如A260/A280
的比值,用于评估样品的纯度,因为蛋白的吸收峰是280nm.纯净的样品,比值大于1.8(DNA)
或者2.0(RNA)。
如果比值低于1.8或者2.0,表示存在蛋白质或者酚类物质的影响。
A230表示样品中存在一些污染物,如碳水化合物,多肽,苯酚等,较纯净的核酸A260/A230的比
值大于2.0。
A320检测溶液的混浊度和其他干扰因子。
纯样品,A320一般是0。
)
三、实验材料与仪器
1、实验材料
pEGFP-N3和pET-28aDNA
2.使用仪器
Eppendorf核酸蛋白测定仪,移液枪
四、实验步骤
1.按下Eppendorf核酸蛋白测定仪dsDNA,比色皿中加入100μlddH2O,blank空白对照。
2.取一0.5ml离心管,吸取质粒DNA5μl,然后再加入95μlddH2O,混匀。
3.将100μl的溶液转移到比色皿中,注意不要出现气泡。
4.按下sample,记录260nm下DNA的浓度(mg/ml)。
并同时记录OD260nm/280nm,
OD260nm/230nm的比值,估测DNA的纯度。
5.计算质粒DNA母液的浓度=OD260nm下的浓度×20(mg/ml),
DNA的纯度:
OD260nm/280nm=1.8±0.1(如果低于1.7,说明样品中蛋白质去除的不完全,或样品中有苯酚的污染;如果高于1.9,说明样品中RNA去除的不完全。
)
正常OD260nm/230nm约为2.5,OD260nm/230nm小于2.0,表示样品中存在一些污染物,如碳水化合物,多肽,苯酚等盐和小分子。
实验三琼脂糖凝胶电泳
一、实验目的
学习掌握一种最常用的分离、鉴定、纯化DNA片段的比较方便、省时的技术:
琼脂糖凝胶电泳的基本原理和操作方法。
二、基本原理
影响DNA在琼脂糖凝胶中迁移速率的因素主要有:
(1)DNA分子的大小双链DNA分子在凝胶基质中迁移的速率与其碱基对数的常用对数成反比。
分子越大,迁移的越慢,因为摩擦阻力越大,也因为大分子通过凝胶孔径的效率低于较小的分子。
(2)琼脂糖浓度给定大小的线
状DNA片段在不同浓度的琼脂糖凝胶中迁移速率不同。
在DNA电泳迁移速率的对数和凝胶浓度
之间存在线性相关。
(3)DNA的构象超螺旋环状(Ⅰ型)、切口环状(Ⅱ型)和线状(Ⅲ型)DNA在琼脂糖凝胶中以不同速率迁移。
其相对迁移速率主要取决于琼脂糖凝胶的浓度和类型,其次是电流强度、缓冲液离子强度和Ⅰ型超螺旋绞紧的程度或密度。
一些条件下,Ⅰ型DNA比
Ⅲ型迁移得快;在另一些条件下,顺序可能相反。
(4)所用的电压低电压时,DNA片段迁移
率与所用的电压成正比。
电场强度升高时,高分子量片段的迁移率遂不成比例的增加。
所以,当电压增大时琼脂糖凝胶分离的有效范围反而减小。
要获得大于2kbDNA片段的良好分辨率,
所用电压不应高于5-8V/cm。
(5)电泳缓冲液DNA的泳动受电泳缓冲液的组成和离子强度的影响。
缺乏离子则电导率降低,DNA或者不动或者迁移很慢。
高离子强度时(如10×buffer),电
导率升高,使得应用适中的电压也会产生大量的热能,最严重时凝胶会熔化,DNA变性。
三、实验材料、仪器及试剂
1、实验材料
质粒DNA
2、使用仪器
核酸电泳仪,小型混合器,冰箱,蓝盾可见光透射仪
四、实验步骤
1、1%琼脂糖凝胶的配制
(1)加20ml1×TBE缓冲液于三角瓶中。
(TBE缓冲液为Tris硼酸-EDTA缓冲溶液,适合长时间电泳,但测得分子质量大于实际分子质量,;TAE缓冲液为Tris醋酸-EDTA缓冲溶液,运用最广泛,较准确但不适合长时间;TPE缓冲液为Tris磷酸-EDTA缓冲溶液,磷酸盐易在乙醇沉淀过程中析出,不适合DNA回
收。
)
(2)精确称取0.2g琼脂糖加到三角瓶中,于微波炉中加热至完全熔化,
(3)冷却至60℃左右,
(4)轻缓倒入封好两端和加上梳子的电泳胶板中,静置冷却30分钟以上,
(5)将胶板除去封胶带,放入电泳缓冲液(TBE)中,使电泳缓冲液刚好没过凝胶约1mm,
轻轻拔除梳子,
(6)取5μl质粒DNA及2μlGenefinder混匀上样。
(也可使用GelRed荧光核酸凝胶染色试剂,用凝胶成像仪显影)
(7)50-100v约电泳0.5-1小时。
(8)蓝盾可见光透射仪观察结果。
五、实验结果
实验四酶切及连接
学习使用限制型内切酶进行DNA酶切的原理和方法。
2.实验原理
迄今已发现了3000多种限制性内切酶。
传统上将限制性内切酶按照亚基组成、酶切位置、识别位点、辅助因子等因素划分为三大类。
II型酶在其识别位点之中或临近的确定位点特异地切开DNA链。
它们产生确定的限制片段,因此是三类限制性内切酶中唯一用于DNA分析和克隆
的一类。
II型限制性内切酶中最普遍的是象EcoRI、HindIII、BamHI和NotI这样在识别序列中进行切割的酶。
这一类酶是构成商业化酶的主要部分。
大部分这类酶都以同二聚体的形式结合
到DNA上,因而识别的是对称序列;但有极少的酶作为单聚体结合到DNA上,识别非对称序列。
一些酶识别连续的序列(如EcoRI识别GAATTC;HindIII识别AAGCTT;BamH识I别G↓GATCC;
NotI识别GC↓GGCCG)C;而另一些识别不连续的序列(如BglI识别GCCNNNNNG)G。
C限制
性内切酶酶切DNA后形成两种类型的末端:
(i)两条链断裂的位置是交错地,产生粘性末端,如
DNA连接酶能够催化在两条DNA链之间形成磷酸二酯键,这种酶需要在一条DNA链的3'-
3.实验材料、仪器及试剂
pEGFP-N3和pET-28aDNA
3.2仪器准备水浴锅、移液器、电泳槽、电泳仪、振荡器、制冰机、蓝盾可见光透射仪、凝胶成像仪
3.3试剂
BamHI(10U/μL)(TaKaRa公司);NotI(10U/μL)(TaKaRa公司),T4ligase
4.操作步骤
4.1酶切
按如下双酶切体系(30μL)混合:
反应物(μL)
pEGFP-N3
pET-28a
质粒
18
18
BamHI
2
2
NotI
2
2
10×bufferK
3
3
0.1%BSA缓冲液
3
3
(BSA缓冲液为牛血清白蛋白,稳定酶的活性。
)
1.离心10s,混匀。
2.37℃水浴酶切3-4h。
3.配制1.0%(M/V)普通琼脂糖凝胶30ml。
(1%(m/v)指:
一般是固体溶于液体的,1g固体溶于100mL水中。
)
4.适当放置冷却,45℃左右倒于电泳胶板上,插好梳子。
5.待凝胶凝固好以后,拨下梳子。
6.酶切样品中加入5μl溴酚蓝-GeneFinder混合液混匀,上样。
7.1×TBEBuffer,80V(3-4V/cm)下电泳30min。
8.荧光激发器观察质粒DNA条带的酶切情况,并照像。
4.2回收酶切产物(采用天为时代DNA回收试剂盒进行回收)
1)配30mL1%进口琼脂糖凝胶,尽量长一些(用粗梳子),对酶切产物进行电泳分离;
2)将酶切产物全部加入加样孔中
3)跑胶,观察结果,并且拍照。
4)用干净的刀片将需要的DNA条带从凝胶上切下来,称取重量。
5)以0.1g凝胶对应300μL的体积加入PN(溶胶液)。
6)50℃水浴放置10min,期间不断温和上下翻动离心管至胶完全融解。
7)
13000rpm离心60s,
将上一步得到的溶液加入到一个吸附柱中,吸附柱再放入收集管,
弃掉废液。
吸附柱,用于吸附层析)
8)加入800μL漂洗液PW,13000rpm离心60s,弃掉废液。
(PW是去盐的洗脱液,目的是洗脱脂类、蛋白及盐类等杂质)
9)加入500μL漂洗液PW,13000rpm离心60s,弃掉废液。
10)将离心吸附柱放回收集管,13000rpm离心2min。
11)取出吸附柱,放入一个干净的离心管中,在吸附膜的中间位置加入适量洗脱缓冲液EB30μL,洗脱缓冲液先在65℃水浴预热,室温放置2min,13000rpm离心1min,然后将离心的溶液重新加回离心吸附柱中,13000rpm离心1min。
12)置于-20℃保存。
按照连接体系进行,16℃连接过夜。
反应物
体积/μL
回收纯化的pET-28a质粒
5
gfp基因片段
12
T4连接酶
1
缓冲液(10×)
2
实验五大肠杆菌感受态细胞的制备及转化
一、实验目的
了解和掌握大肠杆菌感受态细胞的制备方法的原理和操作要点,以及质粒DNA转化大肠杆菌细胞的原理和方法。
二、基本原理
外源DNA只有转化到大肠杆菌细胞内才能得到扩增。
感受态指细菌细胞具有的能够接受外源DNA的一种特殊生理状态。
大肠杆菌的感受态可用CaCl2处理而诱导产生:
将正在生长的大肠杆菌细胞在0℃下加入到低渗的CaCl2溶液中,便会使细胞膜的透性发生改变,此时的细胞即呈现为感受态。
这一方法可以用于批量制备感受态细胞,其转化效率可达到5×106-2×107
个转化克隆子/μg超螺旋质粒DNA。
制备好的大肠杆菌感受态细胞可在-70℃冻存。
在0℃下外源DNA可吸附到感受态细胞表面,短时间的热刺激(42℃,90s)诱导细胞吸
收DNA。
(Ca2+会使细胞膜磷脂双分子层形成液晶结构,促使细胞外膜与内膜间隙中的部分核酸酶解离开来,离开所在区域,诱导细胞成为感受态细胞。
将该体系热激,细胞膜的液晶结构会发生剧烈扰动,并随机出现许多间隙,外源DNA就可能被细胞吸收。
进入细胞的外源DNA分
子通过复制、表达,实现遗传信息的转移,使受体细胞出现新的遗传性状。
)
转化了质粒DNA的大肠杆菌随后在培养基中37℃培养1hr,可使质粒DNA中编码抗生素抗性的基因得以表达,因此,转化了质粒DNA的大肠杆菌细胞可在含有相应抗生素的培养基上
生长,而没有转化的细胞则无法生长。
3.实验材料、仪器1.实验材料
DH5α,BL21,pET-28a重组质粒DNA
2.使用仪器
水浴锅,高压灭菌锅、移液器、超净工作台、离心机、振荡培养箱,制冰机
四、操作步骤
1.LB(Luria-Bertain)液体和固体培养基的配制(参考附录)
氨苄青霉素和卡那霉素等抗生素不抗热,如果培养基温度过高,容易导致抗生素失效,应
使培养基降温至60℃左右后,再加入抗生素。
但也不应使培养基的温度过低,否则容易出现气
泡。
75mm直径的培养皿约需15ml培养基。
2.感受态细胞的制备(CaCl2法)
(1)挑一大肠杆菌单菌落放入3mlLB液体培养基(含Kan+),37℃培养过夜。
(2)活化大肠杆菌:
取3ml新鲜的LB液体培养基加