实时荧光定量PCR技术原理与应用.docx
《实时荧光定量PCR技术原理与应用.docx》由会员分享,可在线阅读,更多相关《实时荧光定量PCR技术原理与应用.docx(9页珍藏版)》请在冰豆网上搜索。
实时荧光定量PCR技术原理与应用
实时荧光定量 PCR技术原理与应用
聚合酶链式反应(PCR)可对特定核苷酸片断进行指数级的扩增。
在扩增反应结束之后,我们可以通过凝胶电泳的方法对扩增产物进行定性的分析,也可以通过放射性核素掺入标记后的光密度扫描来进行定量的分析。
无论定性还是定量分析,分析的都是PCR终产物。
但是在许多情况下,我们所感兴趣的是未经PCR信号放大之前的起始模板量。
例如我们想知道某一转基因动植物转基因的拷贝数或者某一特定基因在特定组织中的表达量。
在这种需求下荧光定量PCR技术应运而生。
所谓的实时荧光定量PCR就是通过对PCR扩增反应中每一个循环产物荧光信号的实时检测从而实现对起始模板定量及定性的分析。
在实时荧光定量PCR反应中,引入了一种荧光化学物质,随着PCR反应的进行,PCR反应产物不断累计,荧光信号强度也等比例增加。
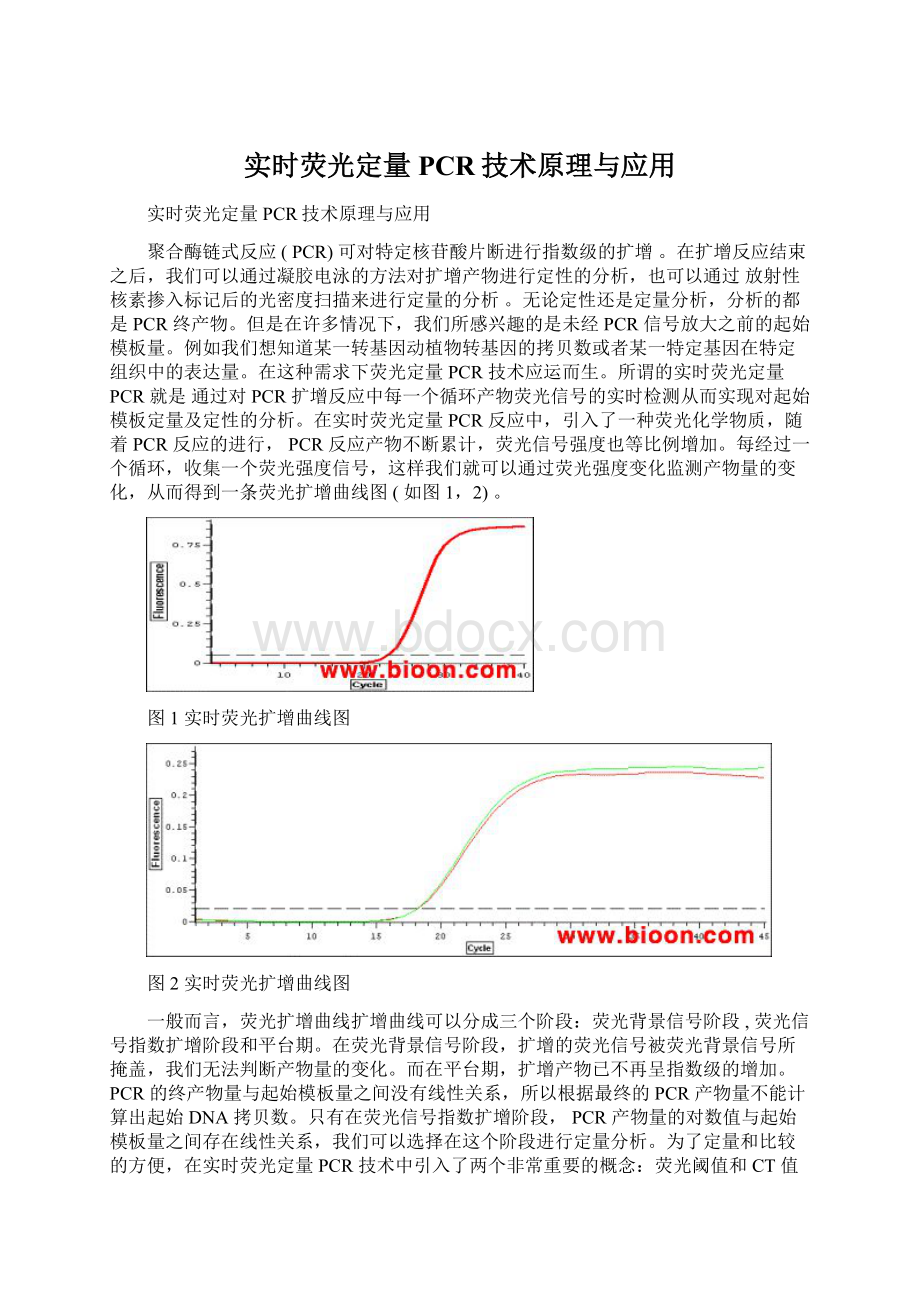
每经过一个循环,收集一个荧光强度信号,这样我们就可以通过荧光强度变化监测产物量的变化,从而得到一条荧光扩增曲线图(如图1,2)。
图1实时荧光扩增曲线图
图2实时荧光扩增曲线图
一般而言,荧光扩增曲线扩增曲线可以分成三个阶段:
荧光背景信号阶段,荧光信号指数扩增阶段和平台期。
在荧光背景信号阶段,扩增的荧光信号被荧光背景信号所掩盖,我们无法判断产物量的变化。
而在平台期,扩增产物已不再呈指数级的增加。
PCR的终产物量与起始模板量之间没有线性关系,所以根据最终的PCR产物量不能计算出起始DNA拷贝数。
只有在荧光信号指数扩增阶段,PCR产物量的对数值与起始模板量之间存在线性关系,我们可以选择在这个阶段进行定量分析。
为了定量和比较的方便,在实时荧光定量PCR技术中引入了两个非常重要的概念:
荧光阈值和CT值。
荧光阈值是在荧光扩增曲线上人为设定的一个值,它可以设定在荧光信号指数扩增阶段任意位置上,但一般我们将荧光域值的缺省设置是3-15个循环的荧光信号的标准偏差的10倍。
每个反应管内的荧光信号到达设定的域值时所经历的循环数被称为CT值(thresholdvalue)。
CT值与起始模板的关系研究表明,每个模板的CT值与该模板的起始拷贝数的对数存在线性关系,起始拷贝数越多,CT值越小。
利用已知起始拷贝数的标准品可作出标准曲线,其中横坐标代表起始拷贝数的对数,纵坐标代表Ct值(如图3所示)。
因此,只要获得未知样品的Ct值,即可从标准曲线上计算出该样品的起始拷贝数。
图2.荧光定量标准曲线
荧光探针和荧光染料
实时荧光定量PCR的化学原理包括探针类和非探针类两种,探针类是利用与靶序列特异杂交的探针来指示扩增产物的增加,非探针类则是利用荧光染料或者特殊设计的引物来指示扩增的增加。
前者由于增加了探针的识别步骤,特异性更高,但后者则简便易行。
1.SYBRGreenI
图4SYBRGREENI工作原理
SYBRGreenI是一种结合于小沟中的双链DNA结合染料。
与双链DNA结合后,其荧光大大增强。
这一性质使其用于扩增产物的检测非常理想。
SYBRGreenI的最大吸收波长约为497nm,发射波长最大约为520nm。
在PCR反应体系中,加入过量SYBR荧光染料,SYBR荧光染料特异性地掺入DNA双链后,发射荧光信号,而不掺入链中的SYBR染料分子不会发射任何荧光信号,从而保证荧光信号的增加与PCR产物的增加完全同步。
SYBRGreenI在核酸的实时检测方面有很多优点,由于它与所有的双链DNA相结合,不必因为模板不同而特别定制,因此设计的程序通用性好,且价格相对较低。
利用荧光染料可以指示双链DNA熔点的性质,通过熔点曲线分析可以识别扩增产物和引物二聚体,因而可以、区分非特异扩增,进一步地还可以实现单色多重测定。
此外,由于一个PCR产物可以与多分子的染料结合,因此SYBRGreenI的灵敏度很高。
但是,由于SYBRGreenI与所有的双链DNA相结合,因此由引物二聚体、单链二级结构以及错误的扩增产物引起的假阳性会影响定量的精确性。
通过测量升高温度后荧光的变化可以帮助降低非特异产物的影响1。
由解链曲线来分析产物的均一性有助于分析由SYBRGreenI得到定量结果。
2.分子信标(molecularbeacon)
分子信标是一种在靶DNA不存在时形成茎环结构的双标记寡核苷酸探针。
在此发夹结构中,位于分子一端的荧光基团与分子另一端的淬灭基团紧紧靠近。
在此结构中,荧光基团被激发后不是产生光子,而是将能量传递给淬灭剂,这一过程称为荧光谐振能量传递(FRET)。
由于"黑色"淬灭剂的存在,由荧光基团产生的能量以红外而不是可见光形式释放出来。
如果第二个荧光基团是淬灭剂,其释放能量的波长与荧光基团的性质有关。
分子信标的茎环结构中,环一般为15-30个核苷酸长,并与目标序列互补;茎一般5-7个核苷酸长,并相互配对形成茎的结构。
荧光基团连接在茎臂的一端,而淬灭剂则连接于另一端。
分子信标必须非常仔细的设计,以致于在复性温度下,模板不存在时形成茎环结构,模板存在时则与模板配对。
与模板配对后,分子信标的构象改变使得荧光基团与淬灭剂分开。
当荧光基团被激发时,它发出自身波长的光子(图5)。
图5.分子信标工作原理
3.TaqMan探针
TaqMan探针是多人拥有的专利技术。
TaqMan探针是一种寡核苷酸探针,它的荧光与目的序列的扩增相关。
它设计为与目标序列上游引物和下游引物之间的序列配对。
荧光基团连接在探针的5’末端,而淬灭剂则在3’末端。
当完整的探针与目标序列配对时,荧光基团发射的荧光因与3’端的淬灭剂接近而被淬灭。
但在进行延伸反应时,聚合酶的5’外切酶活性将探针进行酶切,使得荧光基团与淬灭剂分离。
TaqMan探针适合于各种耐热的聚合酶,如DyNAzymeTMIIDNA聚合酶(MJResearch公司有售)。
随着扩增循环数的增加,释放出来的荧光基团不断积累。
因此荧光强度与扩增产物的数量呈正比关系。
图6.Taqman探针工作原理
4.LUXPrimers
LUX(lightuponextention)引物是利用荧光标记的引物实现定量的一项新技术。
目标特异的引物对中的一个引物3’端用荧光报告基团标记。
在没有单链模板的情况下,该引物自身配对,形成发夹结构,使荧光淬灭。
在没有目标片断的时候,引物与模板配对,发夹结构打开,产生特异的荧光信号(如图7)。
与Taqman探针和分子信标相比,LUX引物通过二级结构实现淬灭,不需要荧光淬灭基团,也不需要设计特异的探针序列。
因为LUX引物是一个相对较新的技术,所以其应用还有待实践的检验。
图7.LUX引物工作原理
实时荧光定量PCR技术的应用
实时荧光定量PCR技术是DNA定量技术的一次飞跃。
运用该项技术,我们可以对DNA、RNA样品进行定量和定性分析。
定量分析包括绝对定量分析和相对定量分析。
前者可以得到某个样本中基因的拷贝数和浓度;后者可以对不同方式处理的两个样本中的基因表达水平进行比较。
除此之外我们还可以对PCR产物或样品进行定性分析:
例如利用熔解曲线分析识别扩增产物和引物二聚体,以区分非特异扩增;利用特异性探针进行基因型分析及SNP检测等。
目前实时荧光PCR技术已经被广泛应用于基础科学研究、临床诊断、疾病研究及药物研发等领域。
其中最主要的应用集中在以下几个方面:
1.DNA或RNA的绝对定量分析。
包括病原微生物或病毒含量的检测,转基因动植物转基因拷贝数的检测,RNAi基因失活率的检测等。
2.基因表达差异分析。
例如比较经过不同处理样本之间特定基因的表达差异(如药物处理、物理处理、化学处理等),特定基因在不同时相的表达差异以及cDNA芯片或差显结果的确证
3.基因分型。
例如SNP检测,甲基化检测等。
随着实时荧光定量PCR技术的推广和普及,该技术必然会得到更广泛的应用。
ProtocolforReal-TimeRT-PCR
Thisprotocoldescribesthedetailedexperimentalprocedureforreal-timeRT-PCRusingSYBRGreenIaPCRprimerbankforquantitativegeneexpressionanalysis.NucleicAcidsResearch31(24):
e154;pp.1-8.PleaserefertothispaperandthePrimerBankHelppageformorebackgroundinformation.ThisprotocolisalsoavailableinMicrosoftWordformathere.Hereisaone-pagesuccinctprotocol.TheprocedurebeginswithreversetranscriptionoftotalRNA.ThecDNAisthenusedastemplateforreal-timePCRwithgenespecificprimers.Youmayneedtomodifythisprotocolifyouusedifferentreagentsorinstrumentsforreal-timePCR.
Timerequired
cDNAsynthesis:
2hours.
real-timePCR:
2hours.
Dissociationcurveanalysis:
0.5hour.
ReagentsandEquipments
OligonucleotidePrimers.GenespecificprimersareretrievedfromPrimerBank(http:
//pga.mgh.harvard.edu/primerbank/).TheseprimersareorderedfromtheMGHDNACorefacility(https:
//dnacore.mgh.harvard.edu/synthesis/index.shtml).AlltheprimersaredesaltedandbothUVabsorbanceandcapillaryelectrophoresisareusedtoassessthequalityofprimersynthesis.
MousetotalliverRNA(Stratagene).
MousetotalRNAmasterpanel(BDBiosciences/Clontech).
SYBRGreenPCRmastermix,200reactions(AppliedBiosystems).
Opticaltubeandcapstrips(AppliedBiosystems).
SuperScriptFirst-StrandSynthesisSystemforRT-PCR(Invitrogen).
25bpDNAladder(Invitrogen).
ABIPrism7000SequenceDetectionSystem(AppliedBiosystems).
ABIPrism7000SDSsoftware(AppliedBiosystems).
3%ReadyAgarosePrecastGel(Bio-Rad).
Agarosegelelectrophoresisapparatus(Bio-Rad).
Detailedprocedure
ReverseTranscription
ReverseTranscriptioniscarriedoutwiththeSuperScriptFirst-StrandSynthesisSystemforRT-PCR.ThefollowingprocedureisbasedonInvitrogen’sprotocol.
1.PreparethefollowingRNA/primermixtureineachtube:
TotalRNA5mg
randomhexamers(50ng/ml)3ml
10mMdNTPmix1ml
DEPCH2Oto10ml
2.Incubatethesamplesat65°Cfor5minandthenoniceforatleast1min.
3.Preparereactionmastermixture.Foreachreaction:
10xRTbuffer2ml
25mMMgCl24ml
0.1MDTT2ml
RNAaseOUT1ml
4.AddthereactionmixturetotheRNA/primermixture,mixbriefly,andthenplaceatroomtemperaturefor2min.
5.Add1ml(50units)ofSuperScriptIIRTtoeachtube,mixandincubateat25°Cfor10min.
6.Incubatethetubesat42°Cfor50min,heatinactivateat70°Cfor15min,andthenchillonice.
7.Add1mlRNaseHandincubateat37°Cfor20min.
8.Storethe1ststrandcDNAat-20°Cuntiluseforreal-timePCR.
Real-timePCR
1.Normalizetheprimerconcentrationsandmixgene-specificforwardandreverseprimerpair.Eachprimer(forwardorreverse)concentrationinthemixtureis5pmol/ml.
2.SetuptheexperimentandthefollowingPCRprogramonABIPrismSDS7000.DonotclickonthedissociationprotocolifyouwanttocheckthePCRresultbyagarosegel.SaveacopyofthesetupfileanddeleteallPCRcycles(usedforlaterdissociationcurveanalysis).Pleasenotetheextensionstepsareslightlydifferentfromdescribedinourpaper.
1)50°C2min,1cycle
2)95°C10min,1cycle
3)95°C15s->60°C30s->72°C30s,40cycles
4)72°C10min,1cycle
3.Areal-timePCRreactionmixturecanbeeither50mlor25ml.Preparethefollowingmixtureineachopticaltube.
25mlSYBRGreenMix(2x)
0.5mllivercDNA
2mlprimerpairmix(5pmol/mleachprimer)
22.5mlH2OOR12.5mlSYBRGreenMix(2x)
0.2mllivercDNA
1mlprimerpairmix(5pmol/mleachprimer)
11.3mlH2O
4.AfterPCRisfinished,removethetubesfromthemachine.ThePCRspecificityisexaminedby3%agarosegelusing5mlfromeachreaction.
5.PutthetubesbackinSDS7000andperformdissociationcurveanalysiswiththesavedcopyofthesetupfile.
6.Analyzethereal-timePCRresultwiththeSDS7000software.Checktoseeifthereisanybimodaldissociationcurveorabnormalamplificationplot.
Troubleshooting
HereIlistedafewmajorcausesforreal-timePCRfailures.PleasereadthePrimerBankHelppageformoredetails.
LittleornoPCRproduct.PoorqualityofPCRtemplates,primers,orreagentsmayleadtoPCRfailures.First,pleaseincludeappropriatePCRcontrolstoeliminatethesepossibilities.Somegenesareexpressedtransientlyoronlyincertaintissues.Inourexperience,thisisthemostlikelycausefornegativePCRresults.Pleasereadliteratureforthegeneexpressionpatterns.Onecaveatisthatmicroarraysarenotalwaysreliableatmeasuringgeneexpressions.Afterswitchingtotheappropriatetemplates,weobtainedpositivePCRresultsincontrasttotheotherwisenegativePCRs(seeourpaperformoredetails).
PoorPCRamplificationefficiency.Theaccuracyofreal-timePCRishighlydependentonPCRefficiency.Areasonableefficiencyshouldbeatleast80%.PoorprimerqualityistheleadingcauseforpoorPCRefficiency.Inthiscase,thePCRamplificationcurveusuallyreachesplateauearlyandthefinalfluorescenceintensityissignificantlylowerthanthatofmostotherPCRs.Thisproblemmaybesolvedwithre-synthesizedprimers.
Primerdimer.Primerdimermaybeoccasionallyobservedifthegeneexpressionlevelisverylow.Ifthisisthecase,increasingthetemplateamountmayhelpeliminatetheprimerdimerformation.
Multiplebandsongelormultiplepeaksinthemeltingcurve.AgarosegelelectrophoresisormeltingcurveanalysismaynotalwaysreliablymeasurePCRspecificity.Fromourexperience,bimodalmeltingcurvesaresometimesobservedforlongamplicons(>200bp)evenwhenthePCRsarespecific.Theobservedheterogeneityinmeltingtemperatureisduetointernalsequenceinhomogeneity(e.g.independentlymeltingblocksofhighandlowGCcontent)ratherthannon-specificamplicon.Ontheotherhand,forshortamplicons(<150bp)veryweak(andfussy)bandsmigratingaheadofthemajorspecificbandsaresometimesobservedonagarosegel.Theseweakbandsaresuper-structuredorsingle-strandedversionofthespecificampliconsinequilibriumstateandthereforeshouldbeconsideredspecific.Althoughgelelectrophoresisormeltingcurveanalysisalonemaynotbe100%reliable,thecombinationofbothcanalwaysrevealPCRspecificityinourexperience.
Non-specificamplicons.