进化树软件MEGA最新606说明书Word文档格式.docx
《进化树软件MEGA最新606说明书Word文档格式.docx》由会员分享,可在线阅读,更多相关《进化树软件MEGA最新606说明书Word文档格式.docx(28页珍藏版)》请在冰豆网上搜索。
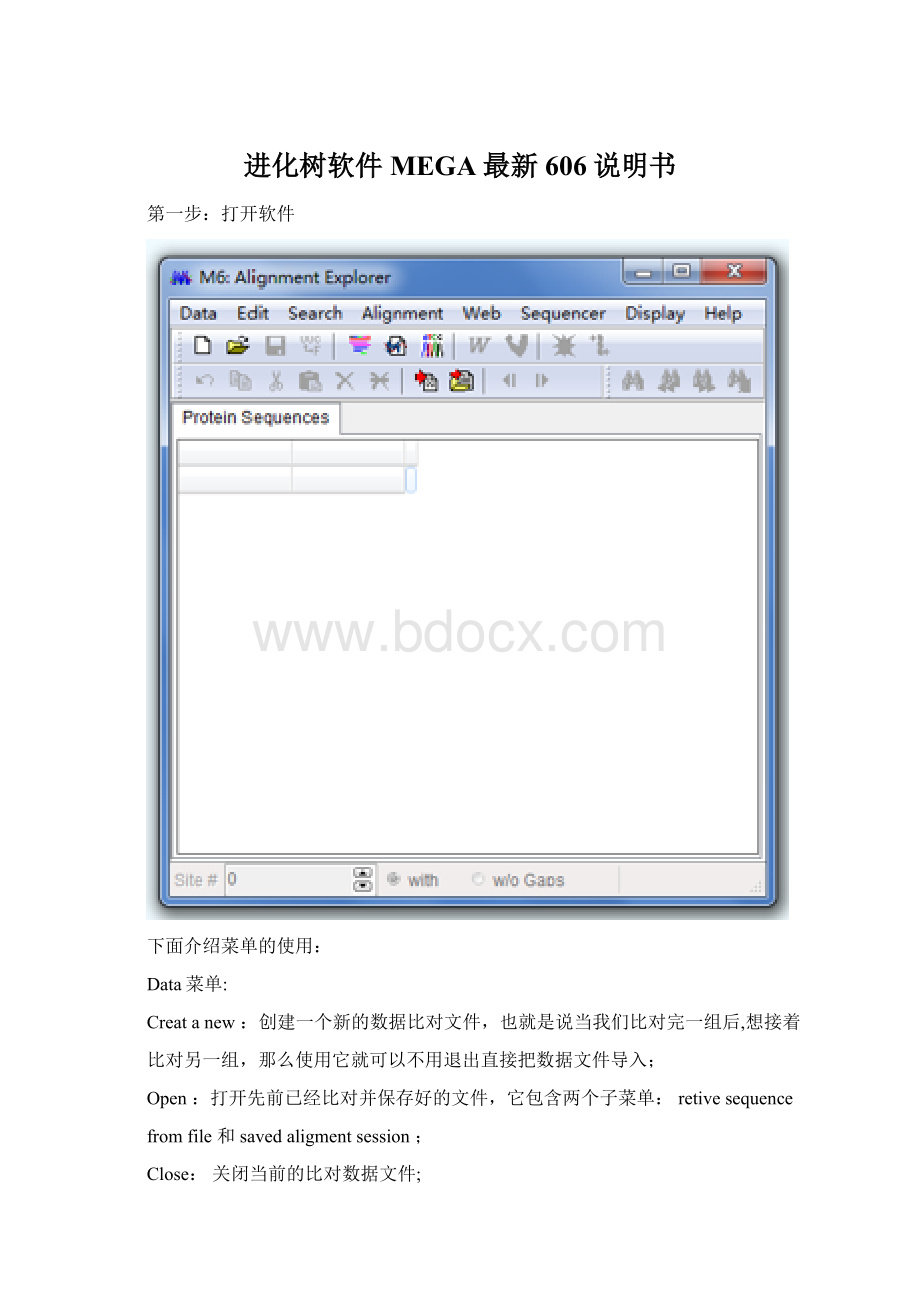
MGTA和FASTA。
DNAsequence:
使用它来选择输入的数据DNA序列,这里需要说明的是如果你输入的
数据是氨基酸序列的话,比对窗口只显示一个标签,若是DNA序列的话则显示两个标
签,一个是DNA序列的,另一个是氨基酸序列的.
Proteinsequences:
选择输入的氨基酸序列,选择后,所以的位点就被当作氨基酸残
基位点来对待.
Translate/untranslate:
只有比对的序列是编码蛋白的DNA序列的时候才可用。
它可以
根据指定的遗传密码表将DNA序列翻译成特定的氨基酸序列。
Selectgeneticcodetable:
使用它将编码蛋白的DNA翻译成特定的蛋白序列。
Reversecomplement:
将选择的一整行的DNA序列变为与之互补配对碱基序列。
Exitalignmentexplorer:
退出序列比对的资源管理窗口
Edit菜单:
使用这个菜单可以对我们的比对序列进行想要的一些编辑工作具体为
Undo:
撤销上一步操作;
Copy:
复制;
Cut:
剪切;
Paste:
粘贴;
这三个操作都可以只针对一个碱基或
氨基酸残基也可以是一段甚至是整个序列;
Delete:
从比对表格中删除一段序列;
Deletegaps:
去掉序列中的空缺;
Insertblanksequence:
重新插入一空行;
标签和序列都是空的;
Insertsequencefromfile:
从已保存的文件中插入新的序列;
Selectsites:
选择
一列序列,与点击比对表上方的灰白空格作用类似;
Selectsequence:
选择一行序列,与点击比对表格左侧的标签名作用类似;
Selectall:
全选;
Allowbaseediting:
只读保护,只有选择后才能对序列进行编辑操作,否则
所以的序列为只读格式,不能进行任何编辑操作。
Search菜单:
用来快捷查找序列中的标记未定或者目的碱基或残基.
Findmotif:
输入你想要查看的一小段序列。
找到后会以黄色标出;
Findnext:
在序列的下游查找目的序列片段;
Findpreious:
在序列的上有查找目的序列片段;
Findmarkedsites:
查找标记位点;
Highlightmotif:
突出标记已经选择的位点。
Web菜单:
这个菜单提供一个链接Genbank的入口,可以在网上直接做Blast搜索。
当
手上没有准备好要比对的序列时,可以直接去网上搜索。
Querygenebanks:
开启NCBI的主页;
Doblastsearch:
开启NCBIBLAST主页;
Showbrowser:
开启网页浏览器。
Sequencer菜单:
此菜单下只有一个子菜单:
editsequencerfile,用来打开一个打开文件对话
框,此对话框可以打开一个sequencerdatafile,一旦打开,这个文件就在trace
datafileviewer/editor的对话框中展示出来。
这个编辑窗口允许你查看和编辑
automatdDNAsequencer产生的tracedata。
它可以阅读和编辑ABI和Staden格式
文件并且序列可以直接被导入到序列比对窗口或被上传到网页浏览器做blast搜
索。
Display菜单:
这个菜单相对简单,主要用来调整工具栏。
Toolbars:
工具栏菜单,它包含一些子菜单,选择后就会出现在比对的窗口
中;
Usecolors:
将不同的位点以不同的颜色显示;
Backgroundcolor:
选择后位点的显示与位点一样的背景颜色;
Font:
字体对话框,通过选择来调整窗口中的序列字符的大小。
Alignment菜单
Mark/unmarksite:
在比对的表格中标记或者不标记一个单一位点,一次每
条序列只能被标记一个位点,不同序列间的位点你可以选择同一列的,也
可以是错开的,要根据自己的目的进行选择。
选择标记后的序列可以使用
alignmarkedsites进行比对分析。
Alignmarkedsites:
比对标记的序列,在这里如果在两个或多个序列间标
记了不在一列的位点重新比对后会出现空格。
Unmarkedallsites:
把所以标记的位点去标记;
Deletegap-onlysite:
去掉序同是空格的一列;
这在多序列比对前很有用。
Auto-fillgaps:
使用空格补齐不同长度的序列.
建树:
1)下载数据
2)初步聚类:
3)建树
进化树的构建另一种方式:
MEGA软件构建系统发育树
摘要:
以白色念珠菌属下面的十个种的18sRNA为例,构建系统发育树来说明MEGA软件的使用方法。
1背景简介
1.1MEGA(分子进化遗传分析)
MEGA的全称是MolecularEvolutionaryGeneticsAnalysis。
MEGAisanintegratedtoolforautomaticandmanualsequencealignment,inferringphylogenetictrees,miningweb—baseddatabases,estimatingratesofmolecularevolution,andtestingevolutionaryhypotheses.MEGA可用于序列比对、进化树的推断、估计分子进化速度、验证进化假说等。
MEGA还可以通过网络(NCBI)进行序列的比对和数据的搜索.
最新版本:
MEGA5.1Beta(软件开发者建议其结果不用于发表文章)
建议下载版本:
MEGA5.05forWindowsandMacOS。
MEGA5hasbeentestedonthefollowingMicrosoftWindows®
operatingsystems:
Windows95/98,NT,2000,XP,Vista,version7,LinuxandMacOS[1]。
MEGA5.05可免费下载,只需输入名字及有效邮箱,下载链接会发送至邮箱,点击可下载.
1.2系统发育树定义
系统发育树(英文:
Phylogenetictree)又称为演化树(evolutionarytree),是表明被认为具有共同祖先的各物种间演化关系的树。
是一种亲缘分支分类方法(cladogram).在树中,每个节点代表其各分支的最近共同祖先,而节点间的线段长度对应演化距离(如估计的演化时间)
1。
3系统发育树的分类
根据有根和无根来区分:
树可分为有根树和无根树两类。
有根树是具有方向的树,根据系统发生树可推断出物种的起源包含唯一的节点,将其作为树中所有物种的最近共同祖先。
最常用的确定树根的方法是使用一个或多个无可争议的同源物种作为外群(英文outgroup),这个外群要足够近,以提供足够的信息,但又不能太近以至于和树中的种类相混。
把有根树去掉根即成为无根树。
一棵无根树在没有其他信息(外群)或假设(如假设最大枝长为根)时不能确定其树根。
无根树是没有方向的,其中线段的两个演化方向都有可能.
基于单个同源基因差异构建的系统发生数应称之为基因树。
因为这种树代表的仅仅是单个基因的进化历史。
而不是它所在物种的进化历史。
物种树一般最好是从多个基因数据的分析中得到。
例如一项关于植物进化的研究中,用了100个不同的基因来构建物种树,因为进化是发生在生物体种群水平上的,而不是发生在个体水平上的,虽然表面上不需要更多的数据,但实际上还是有必要的。
基因树和物种树之间的差异是很重要的,如果只用等位基因来构建物种数,那许多人人和大猩猩就会分到一起,而不是和其他人分到一起.
1.4构建方法
要构建一个进化树(phyligenetictree)。
构建进化树的算法主要分为两类:
独立元素法(discretecharactermethods)和距离依靠法(distancemethods)。
所谓独立元素法是指进化树的拓扑形状是由序列上的每个状态决定的,而距离依靠法是指进化树的拓扑形状由两两序列的进化距离决定的。
进化树枝条的长度代表着进化距离.独立元素法包括最大简约性法(MaximumParsimonymethods)和最大可能性法(MaximumLikelihoodmethods);
距离依靠法包括除权配对法(UPGMAM)和邻位相连法(Neighbor—joining)。
2蛋白质序列分析使用方法
2。
1打开网址http:
//www.ncbi.nlm.nih。
gov/protein/,将菌名输入到protein后面的框内,点Search键,选择一个搜索结果点击进入
2.2将搜索出来的结果选择sendto下拉箭头内的选项,AnalysisTool和BLAST,选择好后点击Submit进行搜索
2.3进入BLAST页面,点击页面最下面的BLAST按钮,进行blast,如图所示:
4从结果中选择10个蛋白质序列,进行复制,粘贴到TXT文档内,然后将TXT文档后缀名改为FASTA
2.5将保存好的,以Fasta做后缀的序列打开
6点击菜单栏内的Alignment选项,选择AlignbyClustalW选项。
7弹出如下图对话框,选择OK键,对数据进行处理
经过一段时间的数据处理,数据处理完成如下图所示:
2.8选择菜单栏中Data选项中的SaveSession选项进行保存.
再选择ExportAlignment中的MEGAFormat和FASTAformat进行保存。
9选择菜单栏中的Analysis选项中的Phylogeny中的Construct/TestMaximumLikelihoodTree选项进行数据处理。
将数据按下表填写,点击Compute键
数据将按下表方式处理:
最大进化树如下所示:
10点击菜单栏下一行的Distance选项,选择下拉菜单中的第一个选项,进行数据处理
出现如下对话框,按下图所示,点击Compute键
即可得出最终结果:
3核酸序列分析使用方法
打开网址http:
//www。
ncbi。
nlm。
nih.gov/nuccore,其余方法同上,如下列图片所示,不再详述.