Vector NTI学习报告.docx
《Vector NTI学习报告.docx》由会员分享,可在线阅读,更多相关《Vector NTI学习报告.docx(20页珍藏版)》请在冰豆网上搜索。
VectorNTI学习报告
VectorNTI学习报告
一,新序列的倒入
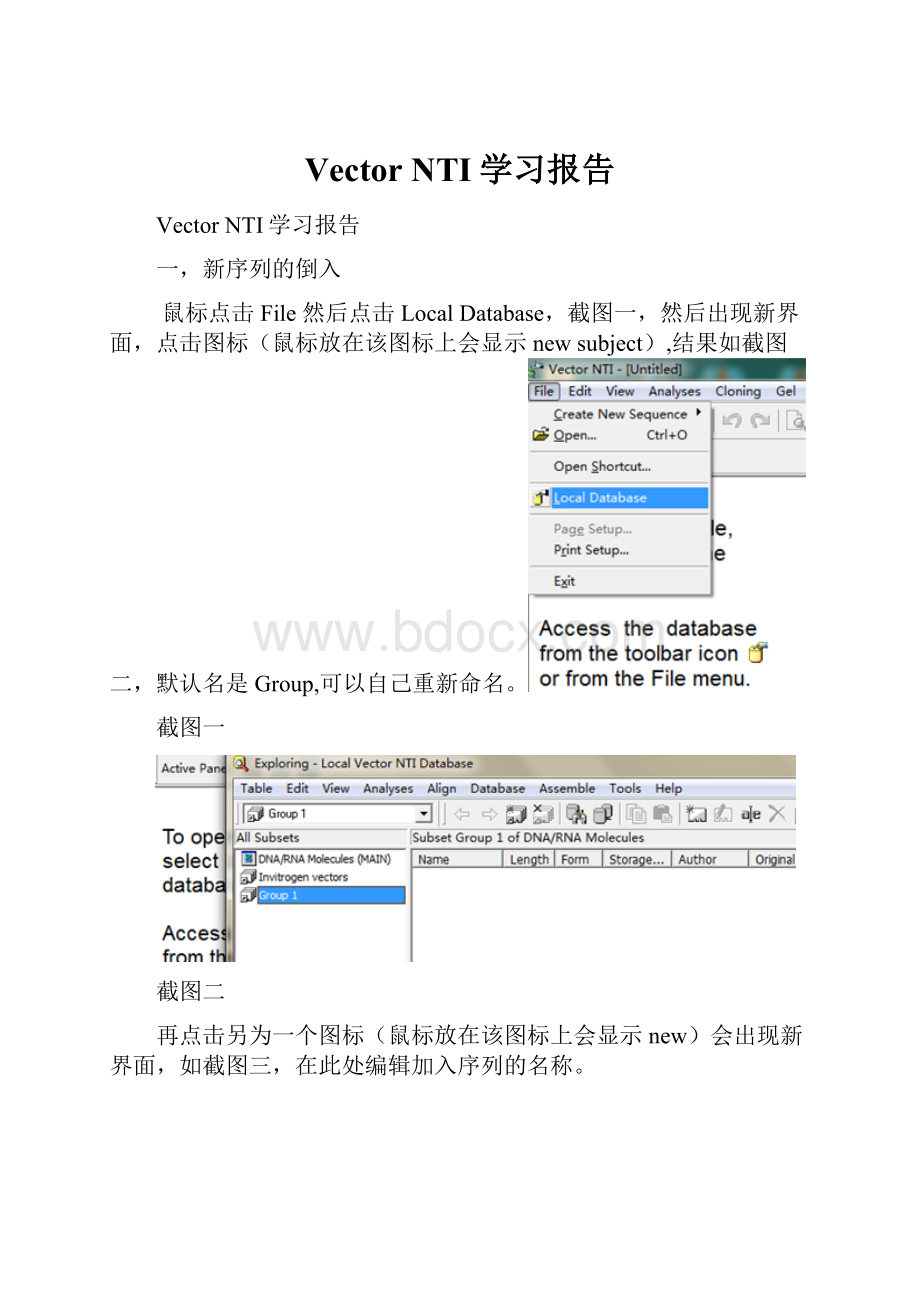
鼠标点击File然后点击LocalDatabase,截图一,然后出现新界面,点击图标(鼠标放在该图标上会显示newsubject),结果如截图二,默认名是Group,可以自己重新命名。
截图一
截图二
再点击另为一个图标(鼠标放在该图标上会显示new)会出现新界面,如截图三,在此处编辑加入序列的名称。
截图三
截图四显示了DNA/RNA的一些特性,根据倒入序列自身的特性选择其中的选项。
截图四
在截图五的界面下(也就是Sequenceandmaps),点击EditeSequence
截图五
出现截图六的窗口
截图六
但是因为我的电脑安装的这个版本不能导入序列,所以点击Paste后,出现了截图七的界面
截图七
二,酶切位点分析
操作过程如截图八,
点击后出现截图九的窗口,这里可以删除和添加想要分析的限制酶。
截图九
我删除了绝大多数限制酶,仅保留三个,见截图十。
截图十
然后我又随机添加了三个,见截图十一,十二,十三
截图十一
截图十二
截图十三
最后点击OK,就可以分析这六个限制酶的酶切位点,见截图十四
截图十四
RFLP可以分析同一个限制酶对多个序列切出的片段数目
具体过程如下,
截图十五
按照截图十五的步骤,会出现如下窗口,
截图十六
我随机选择了两个分子,和一个限制酶,点击calculate
截图十七
结果如下图
截图十八
FindingCommonNon-CuttingEnzyme可以用来分析哪些限制酶不会切割序列
截图十九
按照上图操作,得到下图
截图二十
图二十的左边窗口可以多选,右边窗口也可以多选。
我随机选的几个,然后点击Start,过程与结果见下面两张图
截图二十一
结果可以打印和保存
截图二十二
RestrictionFragments可以分析现在没切的片段大小。
我安装的版本操作不了,所以也没有附上之后会出现的窗口。
截图二十三
截图二十四
RestrictionReport可以对所有限制酶对序列的酶切结果进行统计,操作和结果如下
截图二十五
这份表格可以打印保存
截图二十六
三,功能元件的标记
这个序列是我在软件数据库能随机找的,酶切位点已经被我删去,用来演示标记功能元件的过程。
截图二十七
点击图标
会出现图二十八,左边边是软件自带的图像,每一个图像都有特殊含义,代表特定的生物学功能,右边可以编辑图形的名称和范围
截图二十八
点击OK,结果如下图
截图二十九
如要更改图像显示方式,可点击按钮
会出现下图,选择后,点击OK即可
截图三十
按住Ctrl再点击图标
可以调整图像和文字
截图三十一
还可以点击图标
,就可以对图谱窗口内的图像和文字进行编辑
截图三十二
点击图像可以上下移动,改变形状
截图三十三
点击鼠标右键可以在Properties中进行编辑,见图三十四到三十六
截图三十四
截图三十五
截图三十六
文字也可以通过Properties选项进行编辑,如图三十七到四十
截图三十七
截图三十八
截图三十九
截图四十
点击回形针按钮可以进行批注,如下两图
截图四十一
截图四十二